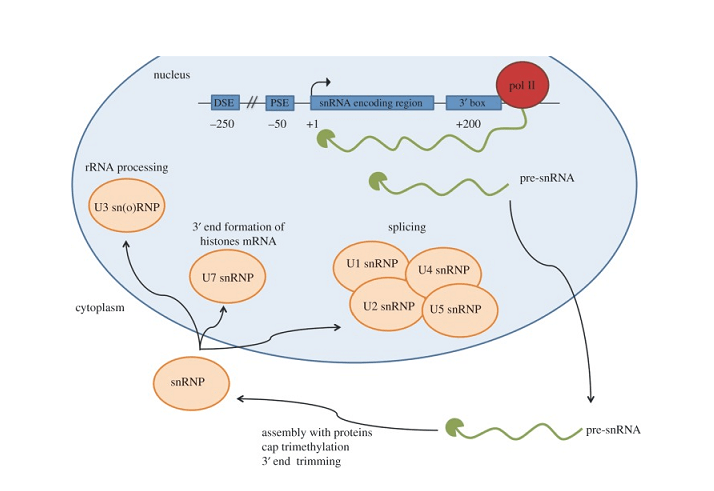
Одиночные ядерные РНК, или snrnas, играть решающую роль в клетках, несмотря на их короткие длины. Как некодирующие молекулы РНК, обнаруженные исключительно в ядре, SNRNAs участвуют в основных посттранскрипционных модификациях, которые помогают синтезировать функциональные белки. Есть два основных класса SNRNAs — U1, U2, U4, U5, и U6, которые образуют мелкие ядерные рибонуклеопротеины (snrnps) вовлечен в сплайсинг, и H/ACA и C/D Snornas, которые направляют химические модификации других RNAS.
Что такое секвенирование РНК с одним нуклеусом (Snrna-seq)?
Секвенирование РНК с одним нуклеусом, или snrna-seq, является инновационным методом, который анализирует ядерный транскриптом, выделенный из отдельных клеток. Этот подход помогает минимизировать транскрипционные реакции на стресс, которые могут искать результаты во время диссоциации. По сравнению с традиционным секвенированием одноклеточной РНК (Scrna-seq), SnRNA-seq демонстрирует определенные преимущества, которые повышают его полезность для изучения клеточной гетерогенности.
В чем разница между snrna-seq и scrna-seq?
Секвенирование РНК с одним нуклеусом (Snrna-seq), также известен как snrna-seq или snuc-seq, Инновационный метод секвенирования РНК, используемый для профилирования экспрессии генов в клетках, которые сложны для изоляции. Этот метод служит мощной альтернативой одноклеточному секвенированию РНК (Scrna-seq) анализируя ядра вместо интактных клеток. Но то, что именно отличает SnRNA-seq от SCRNA-seq? Давайте углубимся в ключевые различия.
Scrna-seq измеряет как цитоплазматические, так и ядерные транскрипты, Предоставление всестороннего взгляда на профиль экспрессии генов клетки. В отличие, SnRNA-seq главным образом измеряет ядерные транскрипты, Поскольку он фокусируется на анализе РНК в самом ядре. Этот целевой подход сводит к минимуму возникновение экспрессии генов, В качестве зрелых рибосомов находятся в цитоплазме. Поэтому, Любые факторы транскрипции, выраженные после процесса диссоциации, не могут быть переведены, Предотвращение транскрипции их нижестоящих целей.
Как SnRNA-seq вмещает сложные типы тканей?
Для одного, SnRNA-seq вмещает более широкий спектр типов тканей, которые могут быть сложными, чтобы полностью диссоциировать на отдельные клетки. Сложные ткани с плотными внеклеточными матрицами, такими как мозг, почка, и сердце может быть проанализировано более эффективно с использованием snRNA-seq из-за его более мягкого протокола диссоциации. Это сохраняет целостность чувствительных типов клеток, которые могут подвергаться транскрипционным изменениям или разлагаться во время более жесткой диссоциации, необходимой для SCRNA-seq.
Каковы преимущества использования snRNA-seq для архивных образцов?
SnRNA-seq также поддерживает изучение архивированных клинических образцов. Замороженные или фиксированные ткани, которые могут быть несовместимы с Scrna-seq, могут быть профилированы до тех пор, пока могут быть изолированы неповрежденные ядра. Это расширяет объем возможных исследований по сравнению с другими методами секвенирования, которые требуют свежеизолированных отдельных клеток. Примечательно, SnRNA-seq позволила исследователям проанализировать посмертную ткань головного мозга человека, чтобы раскрыть разнообразие нейронов на одноклеточном уровне.
Какая уникальная перспектива предоставляет SnRNA-seq?
А не полное представление о полном транскриптоме, SnRNA-seq предоставляет сфокусированную информацию о ядерных процессах и взаимодействиях, модулированных SNRNAS. Он предлагает уникально увеличенный взгляд на регуляторные механизмы в пределах ограниченного ядерного пространства. Применение snRNA-seq к сложным образцам ткани выявило новые типы клеток и состояния, которые могут оставаться незамеченными, используя только Scrna-seq. Такие открытия углубляют наше понимание сотовой организации и специализации, Расширение границ точной медицины.
Как SnRNA-seq может помочь профилировать экспрессию генов в сложных образцах?
Возможности профилирования ядерного профилирования SnRNA-seq оказываются особенно ценными для образцов, которые проблематично анализировать с использованием обычных подходов Scrna-seq. Определенные типы тканей создают технические трудности из -за таких качеств, как плотная матричная состав, хрупкость при диссоциации, или несовместимость с ферментативными процедурами, необходимыми для изоляции одноклеточных. Архивированные образцы, сохранившиеся с помощью фиксации, также представляют проблемы.
Как SnRNA-seq использовалась для изучения сердечной ткани млекопитающих?
Ткань сердца млекопитающего иллюстрирует систему, неразрешимая для Scrna-seq, но поддается анализу SnRNA-seq. Клетки сердца мышц образуют сложную взаимосвязанную сеть, нелегко разрушать без повреждения клеток. Однако, в 2020 Немецкие исследователи успешно секвенировали первое сердце взрослого млекопитающего, используя snrna-seq. Изолируя ядра вместо целых кардиомиоцитов, Они получили практические распределения отличительных клеточных популяций в миокарде. Такое профилирование в масштабе было бы непрактичным с помощью методологии Scrna-seq.
Как SnRNA-seq приносит пользу изучению нейрональных популяций?
Нейрональные популяции представляют собой еще один класс, получающий выгоду от возможностей snRNA-seq. Мозг состоит из неразрывных нейронов, не легко диссоциированных как интактные отдельные клетки. Попытка сделать это может вызвать ответы на стресс, искажая результаты. Тем не менее, изучение транскриптомной неоднородности нейронных подтипов информирует наше понимание нейробиологии, Механизмы болезней, и более. Протоколы snRNA-seq, оптимизированные для ткани ЦНС.
Как SnRNA-seq позволяет анализировать замороженные клинические образцы?
Замороженные клинические образцы аналогично создают ограничения, преодолеваемые SnRNA-seq. Исследования заболеваний печени, сравнивающие здоровые и фиброзные ядра легких, обнаруженные с помощью SnRNA-seq, выявили непредвзятую идентификацию компонентных типов клеток. Такие анализы архивированных биопсиминов пациентов были ранее исключены из анализа из-за входных ограничений SCRNA-seq. Теперь исследователи могут применить ‘Omics Insights к более широкому диапазону реальных образцов, влияющих на точную диагностику и терапию.
Как протокол snRNA-seq сводит к минимуму транскрипционные возмущения?
Основное преимущество SnRNA-seq заключается в его более мягких протоколах диссоциации, которые предотвращают технические проблемы, возникающие в результате жары, длительное время инкубации или чрезмерное ферментативное лечение. КРИБКА, Способный метод лизиса, используемый для ядерного разделения. Не подвергая неповрежденных клеток суровым этапам диссоциации, SnRNA-seq Smpling лучше сохраняет целостность транскриптов, присутствующих во время диссоциации.
Как SnRNA-seq сводит к минимуму ложные паттерны экспрессии генов?
Это сводит к минимуму возникновение лобных паттернов экспрессии генов, вызванных самим процессом диссоциации. Зрелые рибосомы обнаружены исключительно в цитоплазматических компартментах, что означает любые мРНК, кодирующие немедленные гены реакции стресса, экспрессируемые после рассеяния не могут быть переведены. Таким образом, их нисходящие транскрипционные цели остаются неизменными. Сравнительные исследования, применяющие оба метода к одной и той же ткани, показали, что SnRNA-seq захватывает более широкое подмножество типов клеток с меньшим индуцированным диссоциацией транскрипционного возмущения.
Как короткие продолжительности лизиса помогают контролировать транскриптомные смещения в SnRNA-seq?
Дополнительные факторы, такие как короткие продолжительности лизиса, помогают контролировать транскриптомные смещения. Длительное воздействие на лед, прежде чем продолжить приготовление одноназлувшей библиотеки может негативно повлиять на качество данных путем инициируя изменения транскрипции. Тщательное время смягчений таких последствий. Достаточное начальное количество тканей также гарантирует высококачественные препараты ядер, необходимые для надежного профилирования..
Как оптимизированные рабочие процессы snRNA-seq минимизируют технические артефакты?
Общий, Оптимизированные рабочие процессы snRNA-seq минимизируют преаналитические факторы, усложняющие биологическую интерпретацию. Их мягкая извлечение ядерного содержания представляет профили экспрессии генов, формируемых меньше технических артефактов, чем эндогенные биологические процессы. Это придает большую ясность и уверенность в открытиях, полученных в отношении регулирующих сетей, Редкие субпопуляции, и программы специализации клеток в сложных системах in vivo.
Как исследователи могут применить SnRNA-seq для продвижения биологического понимания?
Как инновационная техника, уточняя наш взгляд на клеточную сложность, SnRNA-seq открывает новые границы для различных исследований. Несколько примеров подчеркивают его потенциал для обучения научного прогресса:
- Биология развития: Сравнение ядер с различных эмбриональных стадий может выявить специфичные для стадий регуляторные программы, направляющие дифференциацию. Пространственно разрешенное градиенты развития SNRNA-seq..
- Патология болезней: Профилирование больных по сравнению с здоровыми ядрами ткани может идентифицировать, связанные с патологией изменений в редких типах клеток, освещающие механизмы заболевания. Анализ выборки пациента может проинформировать о том, что яточная диагностика и терапии.
- Нейропсихиатрия: Исследование архивированной посмертной ткани головного мозга через SnRNA-seq помогает охарактеризовать нейродионерность психиатрических состояний. Продольные исследования могут раскрыть продольные биомаркеры и терапевтические цели.
- Эволюционные исследования: SnRNA-seq делает возможные межвидовые сравнения, ранее сложные из-за различий в межорганизмах в свойствах ткани. Такой «Phylo-snrna-seq» информирует наше понимание консервативных и дивергентных регуляторных процессов.
- Мультисомическая интеграция: Сопряжение SnRNA-seq с методами пространственного профилирования дает пространственно разрешенные эпигеномные и транскриптомные карты, выясняющие ядерные микроокружения. Интеграция данных пространственных транскриптов с протеомными или метаболомическими слоями обеспечивает контекст системного уровня.
Краткое содержание
Как один из нескольких новых методов, революционизируя одноклеточный анализ, SnRNA-seq катализирует открытия, делая ранее неразрешимые образцы экспериментально. Его масштабирование ядерной омики обещает свежие идеи, продвигающие различные области от регенеративной медицины до точной онкологии. Вместе с дополнительными подходами, Snrna-seq помогает разгадать увлекательную сложность жизни на беспрецедентном уровне.