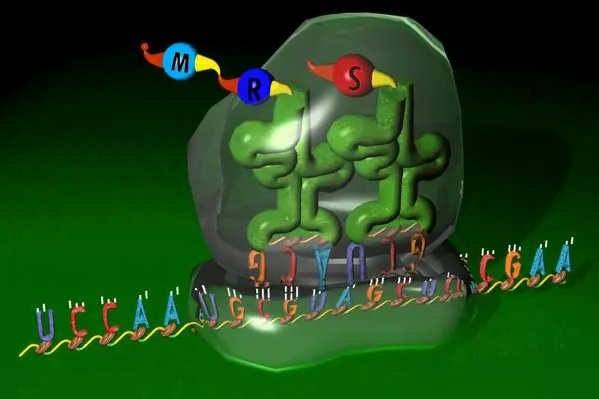
ARN ribosomique, également connu sous le nom d'ARNr, constitue 50% de la teneur totale d'ARN dans une cellule typique et forme un composant clé des ribosomes. Il existe dans trois types principaux – 5S, 5.8S, 16S, et 28s ARNr dans les eucaryotes ou 5s, 16S, et 23S ARNr dans les procaryotes. Avec ribosomal et autres protéines associées, Ces molécules d'ARNr se replient et s'assemblent dans les structures caractéristiques des petites et grandes sous-unités ribosomales.
En plus de fournir une grande partie de l'échafaudage physique pour l'architecture ribosome, Les régions fonctionnelles distinctes au sein des diverses molécules d'ARNr facilitent directement les processus de traduction. Les ARNrs 16S et 23S se situent dans la petite sous-unité des années 30 où ils reconnaissent les codons d'initiation sur l'ARNm et recrutent d'autres facteurs de traduction. Leurs séquences conservées forment des poches moléculaires qui associent précisément les nucléotides messager pour le décodage initial des messages génétiques.
Entre-temps, Les centres catalytiques dans les ARNrs 23 et 28S dans la grande sous-unité des années 60 conduisent la formation de liaisons peptidiques entre les acides aminés entrants. À travers un ensemble d'attaques nucléophiles coordonnées et de transferts de protons, Ces domaines d'ARNr fonctionnels rejoignent le groupe carboxyle d'un acide aminé au groupe amino d'un autre, polymérisation de nouvelles protéines. D'autres régions de l'ARNr aident à stabiliser les intermédiaires d'état de transition et les ARN de transfert aminoacylé (ARNt) pendant l'allongement.
Quel rôle joue l'ARNr dans les ribosomes?
ARN ribosomique, Aussi appelé RRNA, se rattraper 60% du ribosome total. Il remplit à la fois une fonction structurelle et catalytique. Les différentes molécules d'ARNr fournissent l'échafaudage physique qui maintient les deux sous-unités ribosomes ensemble.
En plus, Les régions spécifiques de l'ARNr sont enzymatiquement actives et facilitent directement les réactions impliquées dans la liaison des acides aminés ensemble pendant l'assemblage des protéines. Sans ARNr, Le ribosome n'aurait pas l'architecture 3D appropriée ou les capacités biochimiques pour exercer ses fonctions de production de protéines.
Comment l'initiation de la traduction se produit-elle sur les ribosomes?
La première étape majeure de la synthèse des protéines est l'initiation de la traduction. Cela implique la petite liaison de la sous-unité ribosomale au codon de départ de l'ARN messager (ARNm) avec l'aide de facteurs d'initiation.
L'ARNr 16S dans la petite sous-unité reconnaît et se marie avec la séquence Shine-Dalgarno sur l'ARNm, lui permettant de glisser en position. Ce ARNr 16S agit comme un lecteur de codes à barres moléculaire pour localiser avec précision le site d'initiation.
Que se passe-t-il ensuite dans le processus de traduction?
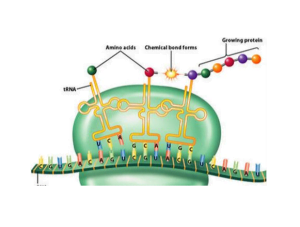
Une fois l'initiation de traduction terminée, L'allongement peut commencer. La grande sous-unité ribosomale rejoint la petite avec l'aide de facteurs d'allongement.
Transférer l'ARN (TRNA) Les molécules transportant des acides aminés activées sont appariées aux codons d'ARNm. L'ARNr 23S dans la grande sous-unité catalyse la formation de liaisons peptidiques entre l'acide aminé entrant et la chaîne de polypeptide en croissance.
Ce processus d'aminoacylation, sélection, La formation d'hébergement et de liaison peptidique se répète le long du modèle d'ARNm jusqu'à ce qu'un codon d'arrêt soit atteint et que la terminaison de traduction se produit.
Comment les différents types d'ARNr sont-ils caractérisés?
Tous les ribosomes contiennent des types de molécules d'ARNr distincts qui adoptent des structures d'ordre supérieur caractéristiques et s'organisent précisément au sein des sous-unités pour remplir des fonctions non redondantes. Dans la plupart des bactéries, Ceux-ci se composent de 5s, 16Les ARNrs S et 23s tandis que les Eucaryotes ont également des variétés de 5,8 et 28. Des analyses biochimiques ont révélé des propriétés uniques de chaque:
5ARNr S – UN 120 ARN nucléotidique qui lie les protéines ribosomales et se replie en un faisceau compact à 5 hélices qui se fait dans la région de protubérance centrale de la sous-unité des années 50. Fournit un échafaudage et aide à coordonner les mouvements intersubitaires.
16ARNr S – À ~ 1500 nucléotides, il forme le noyau structurel de la sous-unité des années 30. Contient quatre domaines distincts qui se plient en éléments de structure secondaire et ponts interdomains critiques pour les interactions de l'ARNm et de l'ARNt.
23ARNr S – Le plus grand ARNr à ~ 2900 nucléotides. Dans la sous-unité des années 50, Il adopte une forme L globale caractéristique avec trois domaines contenant de nombreuses séquences nucléotidiques hautement conservées. Ces poches d'acide nucléique adoptent des conformations adaptées pour catalyser la formation de liaisons peptidiques.
5.8SRNA S et 5S – Résider dans la sous-unité des années 60. 5.8SRNA Spume une structure en épingle à cheveux en épingle à cheveux et un assemblage d'aides. Le ARNr 5S, Bien que plus petit, est de même structuré.
28ARNr S – Équivalent eucaryote de l'ARNr 23S qui contribue au site actif. Une expansion supplémentaire abrite des machines de transfert de peptides supplémentaires et des éléments réglementaires.
Combinant structurel, Des études de mutation et de réticulation ont révélé des emplacements précis de chaque type d'ARNr dans les sous-unités et illustré comment leurs topologies distinctes coopèrent pendant le décodage, relecture et production de protéines.
Comment l'ARNr 16S est-il utilisé en microbiologie?
16Le séquençage du gène de l'ARNr a révolutionné la microbiologie en permettant une identification et une classification microbiennes indépendantes de la culture. Régions de la molécule d'ARNr 16S appelée les régions hypervariables (V1-V9) montrent une variabilité de la séquence nucléotidique entre les organismes mais sont hautement conservés au sein des espèces. Cela fait les régions V idéales moléculaires idéales.
Par RAP Amplifiant l'ADNr 16S d'un échantillon environnemental et déterminant la séquence des régions V3-V5, La signature obtenue peut être exécutée par explosion par rapport aux bases de données de référence des taxons microbiens caractérisés. La base de données frappée avec la correspondance la plus proche indique l'identité phylogénétique du microbe autrefois non cultivé.
Cette technique puissante a aidé à définir l'arbre de vie en regroupant des microbes nouvellement rencontrés en fonction de la parenté évolutive du gène de l'ARNr. Il a permis une détection rapide des agents pathogènes des échantillons cliniques et alimentaires sans cultiver. Les scientifiques l'ont utilisé pour explorer la diversité précédemment cachée dans des environnements exotiques comme le drainage des mines acides ou les évents hydrothermaux.
Plus récemment, Le séquençage d'amplicon d'ARNr à haut débit 16S a révolutionné la recherche sur les microbiomes. Il permet de profilage des communautés microbiennes entières directement à partir de l'ADN microbien dans des échantillons sans biais de culture sélective. La sortie des plates-formes de séquençage de nouvelle génération est traitée à l'aide de pipelines bioinformatiques pour attribuer des séquences taxonomiquement et comparer les assemblages microbiens entre les environnements, niches d'hôte ou groupes de traitement.
Les limitations comprennent l'incapacité de résoudre certaines espèces et les biais d'amorces influençant la couverture. D'autres gènes marqueurs sont maintenant explorés également, Mais l'analyse de l'ADNr 16S reste l'étalon-or en raison de la profondeur de la base de données. Il continue de stimuler d'innombrables découvertes en faisant briller la lumière dans le domaine sombre de la diversité microbienne sur Terre. Les applications vont du diagnostic à la bioprospection et à la compréhension des rôles des microbes dans les écosystèmes, santé, et maladie.
L'ARNr donne-t-il un aperçu des origines de la vie?
Certains scientifiques spéculent l'ARNr peuvent avoir joué un rôle central dans les toutes premières formes de vie sur Terre avant l'émergence de l'ADN et des cellules complexes. En tant que molécule auto-repliante et fonctionnellement active, formant la base de la synthèse des protéines, L'ARNr ou ses ancêtres pourraient représenter un scénario pour la façon dont la vie primitive a évolué son chemin vers une plus grande complexité sur des milliards d'années d'évolution.
Les recherches en cours visent à découvrir des indices en apprenant davantage sur les relations de structure-fonction de l'ARNr à travers divers organismes vivant aujourd'hui et comment ceux qui ont évolué en étape avec des innovations métaboliques et cellulaires du passé antique au présent.
Quelles sont les techniques avancées pour étudier l'ARNr?
Compte tenu de la complexité de l'ARNr, Des méthodes diverses sont utilisées pour éclairer ses myriades de relations structure-fonction à l'échelle variée:
- Le séquençage de nouvelle génération détermine les séquences de gènes d'ARNr à travers les domaines, Aider la taxonomie et la caractérisation des variantes entre les espèces.
- La microscopie cryo-électronique et la cristallographie aux rayons X donnent des structures de résolution presque atomique de ribosomes ou de domaines entiers, révélateur des interactions d'ARNr-RRNA et d'ARNr-protéine.
- Cartes de sondage chimique Implication des nucléotides dans le pliage, Changements et interactions conformationnelles avec les ligands à travers des sites de réactivité aux agents de modification.
- Réticulation couplée à la spectrométrie de masse Pinpoint Régions d'ARNr en contactant des protéines spécifiques ou d'autres biomolécules dans des particules assemblées ou des intermédiaires de réaction.
- FRET à molécule unique surveille le repliement des molécules d'ARNr individuelles et détecte les décalages conformationnels pendant les processus biochimiques comme la translocation.
L'intégration des informations à plusieurs échelles des technologies avancées démêle les relations de structure complexe de l'ARNr, essentielles à la traduction et à l'homéostasie cellulaire avec une profondeur sans précédent. Son importance fondamentale inspire l'innovation continue.
En résumé, tandis que l'ADN est le plus célèbre comme le matériel génétique central de la vie, c'est le héros méconnu – ARNr – Cela garantit l'étape critique de la transformation du code d'ADN en les molécules fonctionnelles qui permettent aux cellules et aux organismes de survivre à travers son rôle essentiel dans la production de protéines via des ribosomes. L'ARNr est vraiment le moteur indispensable de la vie elle-même.