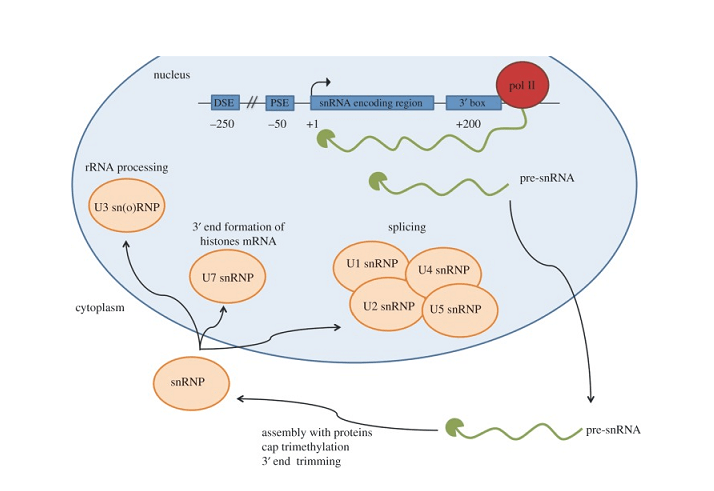
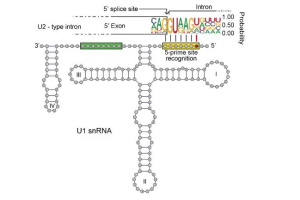
ARN nucléaires simples, ou snRNA, jouent un rôle crucial au sein des cellules malgré leur courte longueur. Comme des molécules d'ARN non codantes trouvées uniquement dans le noyau, Les SNRNA participent à des modifications essentielles post-transcriptionnelles qui aident à synthétiser les protéines fonctionnelles. Il y a deux classes principales de SnRNA – U1, U2, U4, U5, et u6 qui forment les petites ribonucléoprotéines nucléaires (snrnps) impliqué dans l'épissage, et les snornas H / ACA et C / D Box qui guident les modifications chimiques des autres ARN.
Qu'est-ce que le séquençage d'ARN à nucléus unique (snRNA-seq)?
Séquençage de l'ARN à nucléus, ou snrna-seq, est une technique innovante qui analyse le transcriptome nucléaire isolé des cellules individuelles. Cette approche aide à minimiser les réponses du stress transcriptionnel qui peuvent fausser les résultats pendant la dissociation. Par rapport au séquençage traditionnel de l'ARN unique (scrna-seq), SNRNA-Seq présente certains avantages qui améliorent son utilité pour étudier l'hétérogénéité cellulaire.
Quelle est la différence entre SNRNA-Seq et SCRNA-Seq?
Séquençage de l'ARN à nucléus (snRNA-seq), également connu sous le nom de snrna-seq ou snuc-seq, est une méthode innovante de séquençage de l'ARN utilisé pour profiler l'expression des gènes dans les cellules qui sont difficiles à isoler. Cette technique est une alternative puissante au séquençage d'ARN unique (scrna-seq) en analysant les noyaux au lieu de cellules intactes. Mais ce qui distingue exactement SNRNA-SEQ de SCRNA-SEQ? Plongeons dans les principales différences.
SCRNA-Seq mesure les transcriptions cytoplasmiques et nucléaires, Fournir une vue complète du profil d'expression génique d'une cellule. En revanche, SNRNA-SEQ mesure principalement les transcriptions nucléaires, car il se concentre sur l'analyse de l'ARN dans le noyau lui-même. Cette approche ciblée minimise la survenue d'une expression de gènes parasites, Comme résident les ribosomes matures dans le cytoplasme. Donc, Tous les facteurs de transcription exprimés après le processus de dissociation ne peuvent pas être traduits, Empêcher la transcription de leurs cibles en aval.
Comment SnRNA-Seq s'amuse-t-il aux types de tissus difficiles?
Pour un, SNRNA-Seq accueille une plus grande variété de types de tissus qui peuvent être difficiles à se dissocier complètement en cellules uniques. Tissus complexes avec des matrices extracellulaires denses comme le cerveau, rein, et le cœur peut être analysé plus efficacement en utilisant SNRNA-SEQ en raison de son protocole de dissociation plus doux. Cela préserve l'intégrité des types de cellules sensibles qui peuvent subir des changements transcriptionnels ou se dégrader lors de la dissociation plus sévère requise pour SCRNA-SEQ.
Quels sont les avantages de l'utilisation de snrna-seq pour les échantillons archivés?
SNRNA-SEQ soutient également l'étude des échantillons cliniques archivés. Les tissus congelés ou fixes qui seraient incompatibles avec SCRNA-Seq peuvent être profilés tant que les noyaux intacts peuvent être isolés. Cela étend la portée des applications de recherche possibles par rapport à d'autres méthodes de séquençage qui exigent des cellules uniques fraîchement isolées. Notamment, SNRNA-SEQ a permis aux chercheurs d'analyser le tissu cérébral humain post mortem pour découvrir la diversité neuronale au niveau unique.
Quelle perspective unique snrna-seq fournit-il?
Plutôt qu'une vue complète du transcriptome complet, SNRNA-SEQ fournit des informations ciblées sur les processus et les interactions nucléaires modulées par les SNRNA. Il offre une perspective d'amplification unique sur les mécanismes de régulation dans l'espace nucléaire confiné. L'application de SnRNA-Seq à des échantillons de tissus complexes a révélé de nouveaux types de cellules et états qui peuvent être non détectés à l'aide de SCRNA-seq seul. De telles découvertes approfondissent notre compréhension de l'organisation cellulaire et de la spécialisation, Élargir les frontières de la médecine de précision.
Comment SNRNA-SEQ peut-il aider à profiter l'expression des gènes dans des échantillons difficiles?
Les capacités de profilage nucléaire de SNRNA-Seq s'avèrent particulièrement précieuses pour les échantillons qui sont problématiques à analyser en utilisant des approches SCRNA-Seq conventionnelles. Certains types de tissus posent des difficultés techniques en raison de qualités comme la composition de la matrice dense, fragilité lors de la dissociation, ou incompatibilité avec les traitements enzymatiques requis pour l'isolement unique. Des échantillons archivés conservés par la fixation présentent également des défis.
Comment le snrna-seq a-t-il été utilisé pour étudier le tissu cardiaque des mammifères?
Le tissu cardiaque des mammifères illustre un système intraitable à SCRNA-seq mais réalisable à l'analyse SNRNA-Seq. Les cellules musculaires cardiaques forment un réseau interconnecté complexe pas facilement perturbé sans dommage cellulaire. Cependant, dans 2020 Des chercheurs allemands ont réussi à séquencer le premier cœur de mammifères adultes en utilisant SnRNA-Seq. En isolant les noyaux au lieu de cardiomyocytes entiers, Ils ont obtenu des distributions pratiques de populations de cellules distinctives dans le myocarde. Un tel profilage à grande échelle ne serait pas pratique grâce à la méthodologie SCRNA-Seq.
Comment le snrna-seq profite-t-il à l'étude des populations neuronales?
Les populations neuronales représentent une autre classe bénéficiant des capacités de SNRNA-SEQ. Le cerveau se compose de neurones fascinés non facilement dissociés sous forme de cellules uniques intactes. Tenter de le faire peut induire des réponses au stress fausses. Pourtant, étudier l'hétérogénéité transcriptomique des sous-types neuronaux informe notre compréhension de la neurobiologie, mécanismes de maladie, et plus. Les protocoles de SNRNA-SEQ optimisés pour les tissus du SNC conservent des échantillons de cerveau humain archivés et fraîchement réséqués sur une résolution unique de cellules.
Comment SNRNA-Seq permet-il l'analyse des échantillons cliniques gelés?
Les échantillons cliniques congelés posent également les limites surmontées par SnRNA-Seq. Des études de maladie du foie comparant les noyaux pulmonaires sains et fibrotiques détectés via SNRNA-SEQ ont révélé une identification non biaisée des types de cellules constituantes. De telles analyses des biospécimentes archivées des patients ont été précédemment exclues de l'analyse en raison des contraintes d'entrée SCRNA-SEQ. Maintenant, les chercheurs peuvent appliquer «les informations omiques à un plus large éventail d'échantillons du monde réel influençant les diagnostics et la thérapie de précision.
Comment le protocole SNRNA-SEQ minimise-t-il les perturbations transcriptionnelles?
Un avantage majeur de SNRNA-Seq réside dans ses protocoles de dissociation plus douces qui imposent des problèmes techniques résultant de la chaleur, temps d'incubation prolongé ou traitements enzymatiques excessifs. Le bref, La méthode de lyse légère utilisée pour la séparation nucléaire empêche les changements transcriptionnels induits par le stress qui peuvent fausser les résultats SCRNA-SEQ. Sans soumettre des cellules intactes à des étapes de dissociation sévères, L'échantillonnage de SNRNA-SEQ préserve mieux l'intégrité des transcriptions présentes au moment de la dissociation.
Comment SNRNA-SEQ minimise-t-il les modèles d'expression génique parasites?
Cela minimise la survenue de modèles d'expression génique parasites causés par le processus de dissociation lui-même. Les ribosomes matures trouvés uniquement dans les compartiments cytoplasmiques signifient que des ARNm codant pour les gènes de réponse à la contrainte immédiate exprimés après la dissociation ne peuvent pas être traduits. Leurs cibles transcriptionnelles en aval restent donc inchangées. Des études comparatives appliquant les deux techniques aux mêmes tissus ont montré que SNRNA-Seq capture un sous-ensemble plus large de types de cellules avec moins de perturbation transcriptionnelle induite par la dissociation.
Comment les durées de lyse courtes aident-elles à contrôler les biais transcriptomiques dans SNRNA-SEQ?
Des facteurs supplémentaires comme les durées de lyse courtes aident à contrôler les biais transcriptomiques. Une exposition prolongée sur la glace avant de procéder à la préparation de la bibliothèque unique peut avoir un impact négatif sur la qualité des données en initiant les modifications transcriptionnelles. Un timing minutieux apaiser ces effets. Les quantités de tissus initiales suffisantes assurent également les préparations de noyaux de haute qualité nécessaires pour un profilage robuste compte tenu des limitations méthodologiques.
Comment optimiser les workflows SNRNA-SEQ minimiser les artefacts techniques?
Dans l'ensemble, Les workflows SNRNA-SEQ optimisés minimisent les facteurs pré-analytiques compliquant l'interprétation biologique. Leur douce extraction du contenu nucléaire présente des profils d'expression génique en forme moins par des artefacts techniques que les processus biologiques endogènes. Cela donne une plus grande clarté et confiance aux découvertes glanées concernant les réseaux réglementaires, Sous-populations rares, et des programmes de spécialisation cellulaire dans des systèmes complexes in vivo.
Comment les chercheurs peuvent-ils appliquer le SNRNA-SEQ pour faire progresser la compréhension biologique?
En tant que technique innovante, affinant notre vision de la complexité cellulaire, SNRNA-SEQ ouvre de nouvelles frontières pour diverses applications de recherche. Quelques exemples mettent en évidence son potentiel de stimulation scientifique:
- Biologie du développement: La comparaison des noyaux à partir de stades embryonnaires distincts pourrait révéler des programmes de réglementation spécifiques au stade guidant la différenciation. Les gradients de développement de la cartographie de SNRNA-SEQ résolus spatialement peuvent découvrir des mécanismes de motif.
- Pathologie: Profilage malaé par rapport aux noyaux tissulaires sains peut identifier les changements associés à la pathologie dans les types de cellules rares illuminant les mécanismes de maladie. Les analyses des échantillons de patient pourraient éclairer les diagnostics de précision et la thérapie.
- Neuropsychiatrie: L'étude du tissu cérébral archivé post mortem via le snRNA-seq aide à caractériser la neurodiversité des conditions psychiatriques. Des études longitudinales peuvent découvrir des biomarqueurs longitudinaux et des cibles thérapeutiques.
- Études évolutives: SNRNA-Seq fait des comparaisons réticules des espèces intermédiaires auparavant difficiles en raison des différences inter-organismes dans les propriétés tissulaires. Tel “phylo-snrna-seq” informe notre compréhension des processus réglementaires conservés versus divergents.
- Intégration multi-omiques: L'association SNRNA-Seq aux techniques de profilage spatial donne des cartes épigénomiques et transcriptomiques résolues spatialement élucidant les microenvironnements nucléaires. L'intégration des données de transcription spatiale avec des couches protéomiques ou métabolomiques fournit un contexte au niveau des systèmes.
Résumé
Comme l'une des nombreuses techniques émergentes révolutionnantes d'analyse monocellulaire, SNRNA-seq catalyse les découvertes en faisant des échantillons précédemment intraitables expérimentalement traitables. Sa mise à l'échelle des omiques nucléaires promet de nouvelles perspectives avançant divers domaines de la médecine régénérative à l'oncologie de précision. Avec des approches complémentaires, SNRNA-Seq aide à démêler la complexité fascinante de la vie à un niveau sans précédent.