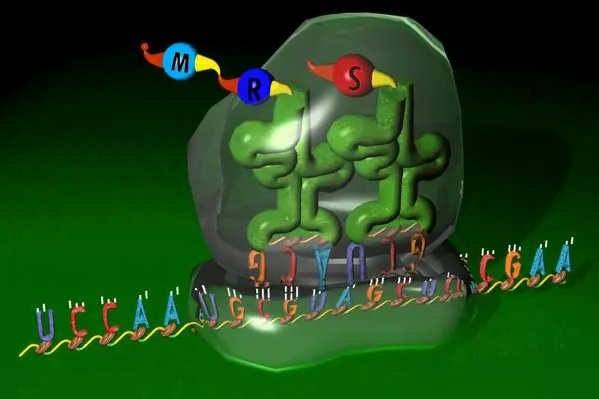
ARN ribosómico, también conocido como rRNA, constituye sobre 50% del contenido total de ARN en una célula típica y forma un componente clave de los ribosomas. Existe en tres tipos principales – 5S, 5.8S, 16S, y 28S rRNA en eucariotas o 5s, 16S, y 23S rRNA en procariotas. Junto con ribosomal y otras proteínas asociadas, Estas moléculas de ARNr se pliozcan y se ensamblan en las estructuras características de las subunidades ribosómicas pequeñas y grandes.
Además de proporcionar gran parte del andamio físico para la arquitectura de ribosoma, Distintas regiones funcionales dentro de las diversas moléculas de rRNA facilitan directamente los procesos centrales de traducción. Los ARNts de los años 16 y 23 se sitúan en la pequeña subunidad de los años 30, donde reconocen los codones de iniciación en el ARNm y reclutan otros factores de traducción. Sus secuencias conservadas forman bolsillos moleculares que combinan precisamente los nucleótidos de mensajeros para la decodificación inicial de los mensajes genéticos.
Mientras tanto, Los centros catalíticos dentro de los ARNm de 23S y 28S en la gran subunidad de los años 60 impulsan la formación de enlaces peptídicos entre los aminoácidos entrantes. A través de un conjunto de ataques nucleofílicos coordinados y transferencias de protones, Estos dominios de ARNr funcionales se unen al grupo carboxilo de un aminoácido al grupo amino de otro, polimerizando nuevas proteínas. Otras regiones de ARNr ayudan a estabilizar los intermedios del estado de transición y los ARN de transferencia aminoacilados (TRNAS) durante el alargamiento.
¿Qué papel juega el rRNA en los ribosomas??
ARN ribosómico, también llamado rRNA, compensar 60% del ribosoma total. Sirve una función estructural y catalítica. Las diversas moléculas de ARNr proporcionan el andamio físico que mantiene unas las dos subunidades de ribosoma.
Además, Las regiones específicas de rRNA son enzimáticamente activas y facilitan directamente las reacciones involucradas en la unión de aminoácidos durante el ensamblaje de proteínas. Sin rRNA, El ribosoma no tendría la arquitectura 3D apropiada o las habilidades bioquímicas para realizar sus deberes de producción de proteínas.
¿Cómo se produce el inicio de la traducción en los ribosomas??
El primer paso importante en la síntesis de proteínas es el inicio de la traducción. Esto implica la pequeña subunidad ribosómica que se une al codón de inicio del ARN mensajero (ARNm) con la ayuda de factores de inicio.
El 16S rRNA dentro de la pequeña subunidad reconoce y se combina con la secuencia Shine-Dalgarno en ARNm, permitiendo que se deslice en posición. Este 16S rRNA actúa como un lector de código de barras molecular para localizar con precisión el sitio de iniciación.
¿Qué sucede después en el proceso de traducción??
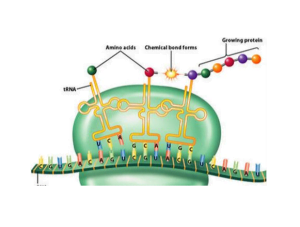
Una vez que se completa la iniciación de la traducción, El alargamiento puede comenzar. La gran subunidad ribosómica se une a la pequeña con la ayuda de factores de alargamiento.
Transferir ARN (tRNA) Las moléculas que transportan aminoácidos activados coinciden con los codones de ARNm. El 23S rRNA en la subunidad grande cataliza la formación de enlaces peptídicos entre el aminoácido entrante y la cadena de polipéptidos en crecimiento.
Este proceso de aminoacilación, selección, La formación de alojamiento y enlace péptido se repite a lo largo de la plantilla de ARNm hasta que se alcanza un codón de parada y se produce la terminación de la traducción.
¿Cómo se caracterizan los diferentes tipos de ARNr??
Todos los ribosomas contienen distintos tipos de moléculas de ARNr que adoptan estructuras características de orden superior y se organizan con precisión dentro de las subunidades para realizar funciones no redundantes. En la mayoría de las bacterias, estos consisten en 5s, 16Sy 23S rRNA, mientras que los eucariotas también tienen variedades 5.8 y 28S. Los análisis bioquímicos han revelado propiedades únicas de cada uno:
5S rRNA – A 120 ARN de nucleótidos que se une a las proteínas ribosómicas y se pliega en un paquete compacto de 5 hélice que se incluye en la región de protuberancia central de la subunidad de los años 50. Proporciona un andamio y ayuda a coordinar los movimientos entre subunidades.
16S rRNA – A ~ 1500 nucleótidos, Forma el núcleo estructural de la subunidad de los años 30. Contiene cuatro dominios distintos que se doblan en elementos de estructura secundaria y puentes interdominio críticos para las interacciones de ARNm y tRNA.
23S rRNA – El rRNA más grande a ~ 2900 nucleótidos. Dentro de la subunidad de los años 50, Adopta una forma de L general característica con tres dominios que contienen muchas secuencias de nucleótidos altamente conservadas. Estos bolsillos de ácido nucleico adoptan conformaciones adecuadas para catalizar la formación de enlaces de péptidos.
5.8Sy 5s rRNA – Residir en la subunidad de los años 60. 5.8S rRNA asume una estructura de cabello de cabello y el ensamblaje de SIDA. El 5s rRNA, Mientras que es más pequeño, está estructurado de manera similar.
28S rRNA – Equivalente eucariota de 23S rRNA que contribuye al sitio activo. La expansión adicional alberga maquinaria de transferencia de péptidos adicionales y elementos reglamentarios.
Combinando estructural, Los estudios mutacionales y de reticulación han revelado ubicaciones precisas de cada tipo de rRNA dentro de las subunidades e ilustran cómo cooperan sus distintas topologías durante la decodificación, Corriente y producción de proteínas.
¿Cómo se usa 16S rRNA en microbiología??
16La secuenciación del gen s rRNA revolucionó la microbiología al permitir la identificación y clasificación microbianas independientes del cultivo. Regiones dentro de la molécula de ARNr 16S llamada regiones hipervariables (V1-V9) Mostrar variabilidad en la secuencia de nucleótidos entre organismos, pero están altamente conservados dentro de las especies. Esto hace que las regiones V regiones sean ideales moleculares.
Por PCR Amplificando 16S rDNA a partir de una muestra ambiental y determinando la secuencia de las regiones V3-V5, La firma obtenida se puede ejecutar a través de BLAST contra bases de datos de referencia de taxones microbianos caracterizados. El golpe de la base de datos con la coincidencia más cercana indica la identidad filogenética del microbio anteriormente no cultivado.
Esta poderosa técnica ayudó a definir el árbol de la vida agrupando microbios recién encontrados basados en la relación evolutiva del gen rRNA. Permitió una rápida detección de patógenos de muestras clínicas y de alimentos sin cultivo.. Los científicos lo usaron para explorar la diversidad previamente oculta en entornos exóticos como el drenaje de minas ácidas o los respiraderos hidrotérmicos.
Más recientemente, secuenciación de amplicón de ARNm de alto rendimiento de alto rendimiento revolucionó la investigación del microbioma. Permite perfilar comunidades microbianas completas directamente del ADN microbiano en muestras sin sesgo de cultivo selectivo. La salida de las plataformas de secuenciación de próxima generación se procesa utilizando tuberías bioinformáticas para asignar secuencias taxonómicamente y comparar ensamblajes microbianos entre entornos, Niches o grupos de tratamiento.
Las limitaciones incluyen la incapacidad de resolver algunos sesgos de especies y cebadores que influyen en la cobertura. Otros genes marcadores ahora también se exploran, Pero el análisis de ADNr 16S sigue siendo el estándar de oro debido a la profundidad de la base de datos. Continúa estimulando innumerables descubrimientos al brillar la luz en el oscuro reino de la diversidad microbiana en la Tierra.. Las aplicaciones van desde el diagnóstico hasta la bioprospección y la comprensión de los roles de los microbios en los ecosistemas, salud, y enfermedad.
¿RRNA proporciona información sobre los orígenes de la vida??
Algunos científicos especulan ARNr pueden haber jugado un papel fundamental en las formas de la primera vida en la Tierra antes de la aparición de ADN y células complejas.. Como una molécula autorreplicante y funcionalmente activa que forma la base de la síntesis de proteínas, rRNA o sus antepasados podrían representar un escenario de cómo la vida primitiva arrugó su camino hacia una mayor complejidad durante miles de millones de años de evolución.
La investigación en curso tiene como objetivo descubrir pistas aprendiendo más sobre las relaciones de estructura de rRNA-función de la estructura en diversos organismos que viven hoy y cómo los evolucionaron con las innovaciones metabólicas y celulares del pasado antiguo hasta el presente.
¿Cuáles son las técnicas avanzadas para estudiar rRNA??
Dada la complejidad de rRNA, Se emplean diversos métodos para iluminar sus innumerables relaciones de estructura de estructura a escalas variadas:
- La secuenciación de próxima generación determina las secuencias de genes de rRNA en los dominios, ayudar a la taxonomía y la caracterización de las variantes entre especies.
- La microscopía crioelectron y la cristalografía de rayos X producen estructuras de resolución casi atómica de ribosomas o dominios enteros, revelando las interacciones rRNA-ARNm y rRNA-proteína.
- Mapas de sondeo químico La participación de los nucleótidos en el plegado, Cambios conformacionales e interacciones con ligandos a través de sitios de reactividad a agentes modificadores.
- La reticulación junto con la espectrometría de masas señala las regiones de ARNr que contactan proteínas específicas u otras biomoléculas dentro de partículas ensambladas o intermedios de reacción.
- El FRET de molécula única monitorea el plegamiento de moléculas de ARNt individuales y detecta cambios conformacionales durante los procesos bioquímicos como la translocación.
La integración de ideas a múltiples escala de tecnologías avanzadas está desentrañando las intrincadas relaciones de estructura de estructura de rRNA esenciales para la traducción y la homeostasis celular con profundidad sin precedentes. Su importancia fundamental inspira la innovación continua.
En resumen, mientras que el ADN es más famoso como el material genético central de la vida, Es el héroe no reconocido – rRNA – Eso asegura el paso crítico de transformar el código de ADN en las moléculas funcionales que permiten que las células y los organismos sobrevivan a través de su papel esencial en la producción de proteínas a través de los ribosomas. rRNA es realmente el motor indispensable de la vida en sí mismo.