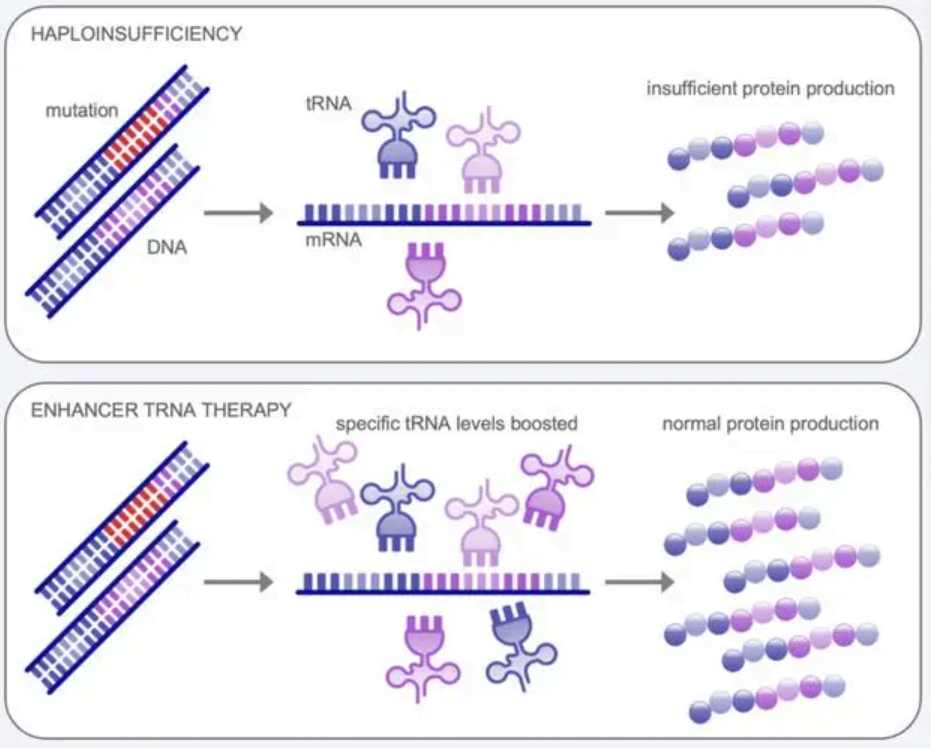
Was ist trna?
RNA übertragen, allgemein bekannt als tRNA, spielt eine wichtige Rolle bei der Proteinsynthese durch Überbrückungscodons in Messenger -RNA (mRNA) mit ihren entsprechenden Aminosäuren. Jedes tRNA -Molekül enthält eine Anticodon -Schleife, die bestimmte Codons durch Basispaarung erkennt.
Dies ermöglicht TRNA, die korrekte Aminosäure zu liefern, die vom mRNA -Codon für die Proteinanordnung dem Ribosom angegeben ist. Es gibt vorbei 500 Verschiedene tRNA -Gene, die im menschlichen Genom gefunden wurden, das TRNAs codiert, die alle tragen 20 Standard -Aminosäuren. Dies stellt sicher, dass genügend TRNAs verfügbar sind, um die effiziente Übersetzung des genetischen Codes zu erleichtern.
Wie erkennt TRNA mRNA -Codons??
Der Schlüssel zur Funktion von TRNA liegt in seinem Anticodon, Eine Sequenz von drei Nukleotiden, die Wasserstoffbrückenbindungen mit einem komplementären mRNA -Codon bilden. Diese Basispaarung folgt Watson-Crick-Komplementaritätsregeln, wo Adenin (A) Paare mit Uracil (U) und Zytosin (C) Paare mit Guanine (G).
Eine gegebene TRNA trägt nur eine Art Aminosäure, bestimmt durch seine spezifische Anticodon -Sequenz. Zum Beispiel, Eine tRNA mit dem Anticodon -UAC würde das Codon Aug erkennen und das Aminosäure -Methionin anbringen. Diese genaue Basispaarung ermöglicht es TRNAs, Codons treu zu übersetzen und die richtige Proteinsequenz zusammenzustellen.
Was ist Wackelpaarung und wie erweitert es den genetischen Code??
Einige TRNAs zeigen ein Phänomen, das als Wobble -Paarung bekannt ist, wobei die dritte Position des Antikodons Basenpaare mit mehr als einem Nukleotid im Codon bilden kann. Zum Beispiel, ein Guanine (G) In der Anticodon kann die dritte Position mit beiden Cytosin kombinieren (C) oder Uracil (U) im mRNA -Codon.
Diese erhöhte Flexibilität an der dritten Anticodon -Position ermöglicht es einer einzelnen TRNA, mehrere Codons zu erkennen, die dieselbe Aminosäure angeben. Durch Wackelpaarung, Eine einzelne tRNA kann an zwei oder mehr Codons binden. Dies verbessert die Translationseffizienz, indem die Anzahl unterschiedlicher tRNA -Moleküle wirtschaftlich zur Übersetzung des genetischen Code.
Wie werden Aminosäuren an TRNAs gebunden??
Aminoacyl-tRNA-Synthetasen katalysieren die Bindung spezifischer Aminosäuren an verwandte tRNA-Moleküle. Jede Synthetase erkennt nur eine tRNA und ihre entsprechende Aminosäure. Die Reaktion wird durch hydrolyse ATP auf AMP und Pyrophosphat angetrieben. Jede Synthetase besitzt unverwechselbare Strukturelemente und Bindungstaschen.
Gelegentlich treten Fehler bei der Bearbeitung von falsch angeschlossenen Aminosäuren auf. Korrekturlesenschritte wie Hydrolyse von falsch aufgeladenen TRNAs schützen vor Fehlern und gewährleisten die Übersetzung des genetischen Code.
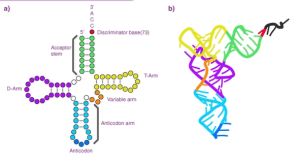
Was ist die Struktur eines tRNA -Moleküls?
Die primäre Sequenz einer tRNA faltet sich zu einer charakteristischen L-förmigen dreidimensionalen Struktur, die durch intra-molekulares Basenpaaring stabilisiert ist. Ein Arm verfügt über die Anticodon -Schleife, während der andere Ende die angebrachte Aminosäure verankert. Verschiedene tRNA-Transkripte, die subtil unterschiedliche Strukturen annehmen.
Diese robuste, aber anpassungsfähige Architektur bietet perfekt tRNA, um Aminosäure -Ladungen und Abgabe an das Ribosom zu unterziehen. Die molekulare Modellierung hat Einblicke in strukturelle Umlagerungen geliefert, die die funktionellen Übergänge der tRNA während der Proteinsynthese begleiten können.
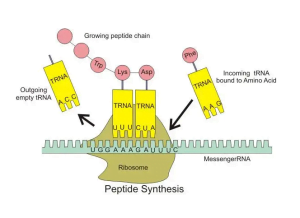
Wie interagieren TRNAs mit dem Ribosom??
Nach der Aminoacylierung, TRNAs interagieren mit mRNA und dem Ribosom, um die Proteinbaugruppe zu erleichtern. Das Ribosom enthält drei Bindungsstellen für TRNAs – die a, P- und E -Websites. Laden Sie geladene TRNAs in die A -Site und Paar mit verwandten Codons. Katalysiert durch Dehnungsfaktoren, Die Bildung von Peptidbindungen verbindet die eingehende Aminosäure mit der wachsenden Polypeptidkette, gefolgt von der Translokation von TRNAs zu den P- und E -Stellen.
Leere TrnAs verlassen das Ribosom vom E -Standort, das für nachfolgende Übersetzungsrunden aufgeladen werden soll. Präzise koordinierte Bewegungen der mRNA, TRNAs und ribosomale Untereinheiten treiben die Proteinproduktionsfabrik an.
Wie werden TRNAs transkribiert??
TRNAs werden aus Genen im Kern durch RNA -Polymerase III transkribiert. TRNA -Gene enthalten eine interne Promotorregion, die durch RNA -Polymerase III und andere Transkriptionsfaktoren erkannt wird. Das anfängliche Transkript ist eine lange Vorläufer -TRNA, die zusätzliche Sequenzen enthält, die verarbeitet werden müssen.
Dieser Vorläufer erfährt 5′ Capping, Intron -Spleißen, und 3′ Anhängerzusatz. Enzyme machen präzise Schnitte, um flankierende Sequenzen abzuschneiden und Exons zu löschen, Nur die kanonische Kleeblatt -Sekundärstruktur verlassen.
Welche Rolle spielen Modifikationsenzyme??
Nach Transkription und Verarbeitung, TRNAs werden umfangreiche posttranskriptionelle Modifikationen unterzogen, die durch verschiedene Modifikationsenzyme katalysiert wurden. Über 100 In TRNAs wurden unterschiedliche Modifikationen gefunden, Die Mehrheit, die an bestimmten Nukleotiden im Anticodon -Stamm und der Schleifenregion auftritt.
Modifikationen Feinstrukturstabilität, Bindende Affinitäten, Erkennungselemente und zelluläre Lokalisierungssignale. Sie spielen eine wichtige Rolle beim Korrekturlesen, Verhinderung von Frameshieren und Erleichterung der Rekrutierung des Dehnungsfaktors während der Übersetzung.
Wie werden TRNAs in das Zytoplasma transportiert??
Die entstehenden TRNAs werden vom Kern zum Zytoplasma zur Verwendung in der Translation exportiert. Der Export wird durch bestimmte Träger und Transporter erleichtert. In Eukaryoten, Viele TRNAs enthalten ein Transportelement, das die Erkennung durch Exportin-T und Bewegung durch nukleare Porenkomplexe ermöglicht. Spezialisierte Transportfaktoren binden zusammen mit Ran-GTP, um TRNAs in das Zytoplasma zu liefern, wo sie sich dem Pool anschließen. Der retrograde nukleare Import von aberranten TRNAs bietet einen Quality Control Checkpoint.
Was ist der Lebenszyklus von TRNAs??
Nach der Aminoacylierung im Zytoplasma, TRNAs assoziieren vorübergehend mit Dehnungsfaktoren und rekrutieren Aminosäuren für die Proteinsynthese am Ribosom. Jede Übersetzungsrunde wird geschätzt, um es zu beteiligen 200 Reaktivzyklen durch ein einzelnes tRNA -Molekül.
Da werden TRNAs kontinuierlich regeneriert, Sie müssen präzise Strukturen aufrechterhalten und dem Verschlechterung widerstehen. Gebrauchte TRNAs werden vom Ribosom zerlegt und repariert, Recycling oder Verschlechterung in Abhängigkeit vom Ausmaß des Schadens. Qualitätskontrolle sorgt nur intakte und voll funktionsfähige TRNAs unterstützen neue Übersetzungsrunden.
Warum ist das Verständnis von TRNA für wissenschaftliche Forschung und menschliche Gesundheit wichtig?
Das Verständnis der vollständigen Funktionen von TRNA ist entscheidend, um unser Wissen über grundlegende biologische Prozesse voranzutreiben und potenzielle therapeutische Interventionen zu entwickeln. Indem Sie die Mechanismen entwirren, durch die tRNA -Moleküle an der Translation teilnehmen, Forscher können Einblicke in die zugrunde liegenden Ursachen genetischer Störungen gewinnen, Krebs, und andere Krankheiten, die mit Fehlern in der Proteinsynthese verbunden sind.
Darüber hinaus, Die Fähigkeit, tRNA -Moleküle zu konstruieren oder zu modifizieren, verspricht für verschiedene Anwendungen, wie die Herstellung biotherapeutischer Proteine mit nicht standardmäßigen Aminosäuren oder Entwicklung neuer antimikrobieller Mittel, die auf die Translationsmaschinerie pathogener Organismen abzielen.
Zusammenfassung
Abschließend, Während der Größe kompakt, TRNAs spielen eine facettenreiche Rolle bei der Genexpression. Ihre Transkription, Verarbeitung, Modifikation und intrazelluläre Verteilung beinhalten eine Reihe von Enzymen und Transportern, die in exquisiter Koordination arbeiten. Die kontinuierliche Regeneration ist von entscheidender Bedeutung, um die hohen Anforderungen der Proteinsynthese aufrechtzuerhalten. Die enge Regulierung bei jedem Schritt schützt die Integrität und Treue des genetischen Code. Ein ausgewogener tRNA -Pool bildet die Grundlage für eine genaue und effiziente Proteinproduktion in allen lebenden Zellen.