Ribosomale RNA, auch als rRNA bekannt, ist vorbei 50% des Gesamt -RNA -Gehalts in einer typischen Zelle und bildet eine Schlüsselkomponente von Ribosomen. Es existiert in drei Haupttypen – 5S, 5.8S, 16S, und 28S -rRNA in Eukaryoten oder 5s, 16S, und 23s rRNA in Prokaryoten. Zusammen mit ribosomalen und anderen assoziierten Proteinen, Diese rRNA.
Neben der Bereitstellung eines Großteils des physischen Gerüsts für die Ribosomenarchitektur, Unterschiedliche funktionelle Regionen innerhalb der verschiedenen rRNA -Moleküle erleichtern direkt Kernprozesse der Translation. Die 16S und 23S RRNAs befinden. Ihre konservierten Sequenzen bilden molekulare Taschen, die genau Messenger -Nukleotide für die anfängliche Dekodierung genetischer Botschaften kombinieren.
In der Zwischenzeit, Katalytische Zentren innerhalb der 23S- und 28S -RRNAs in der großen 60er -Untereinheit treiben die Bildung von Peptidbindungen zwischen eingehenden Aminosäuren an. Durch eine Reihe koordinierter nucleophiler Angriffe und Protonenübertragungen, Diese funktionellen rRNA, Polymerisierende neue Proteine. Andere rRNA -Regionen helfen dabei (trnas) während der Verlängerung.
Welche Rolle spielt rRNA in Ribosomen??
Ribosomale RNA, auch rRNA genannt, Make up herum 60% des gesamten Ribosoms. Es dient sowohl einer strukturellen als auch einer katalytischen Funktion. Die verschiedenen rRNA -Moleküle liefern das physikalische Gerüst, das die beiden Ribosomenuntereinheiten zusammenhält.
Zusätzlich, Spezifische Regionen rRNA sind enzymatisch aktiv und erleichtern die Reaktionen,. Ohne rRNA, Das Ribosom hätte nicht die entsprechende 3D -Architektur oder biochemische Fähigkeiten, um seine Aufgaben zur Proteinproduktion auszuführen.
Wie tritt auf Ribosomen die Übersetzungsinitiierung auf?
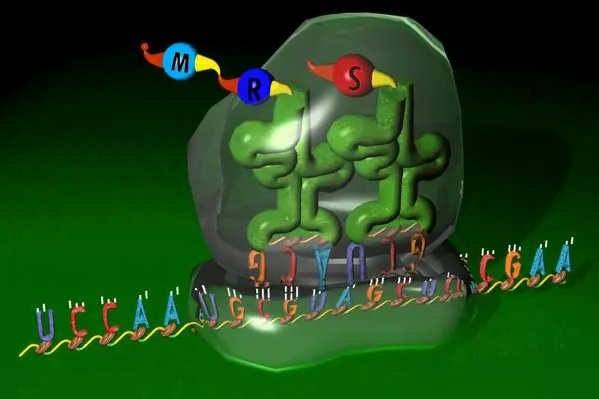
Der erste große Schritt bei der Proteinsynthese ist die Initiierung der Translation. Dies beinhaltet die kleine ribosomale Untereinheit -Bindung an das Startcodon der Messenger -RNA (mRNA) Mit Hilfe von Initiationsfaktoren.
Die 16S-rRNA innerhalb der kleinen Untereinheit erkennt und passt mit der Shine-Dalgarno-Sequenz auf mRNA, lassen, dass es in Position gleiten kann. Diese 16S -rRNA fungiert als molekularer Barcode -Leser, um die Initiationsstelle genau zu lokalisieren.
Was passiert als nächstes im Übersetzungsprozess?
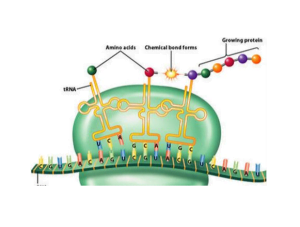
Sobald die Übersetzungsinitiation abgeschlossen ist, Dehnung kann beginnen. Die große ribosomale Untereinheit verbindet sich der kleinen mit Hilfe von Dehnungsfaktoren.
RNA übertragen (tRNA) Moleküle, die aktivierte Aminosäuren tragen. Die 23S -rRNA in der großen Untereinheit katalysiert die Peptidbindungsbildung zwischen der eingehenden Aminosäure und der wachsenden Polypeptidkette.
Dieser Prozess der Aminoacylierung, Auswahl, Die Bildung von Unterbringungs- und Peptidbindungen wiederholt sich entlang der mRNA.
Wie sind verschiedene rRNA -Typen charakterisiert??
Alle Ribosomen enthalten unterschiedliche rRNA-Molekültypen, die charakteristische Strukturen höherer Ordnung anwenden und genau in Untereinheiten organisieren, um nicht redundante Funktionen auszuführen. In den meisten Bakterien, Diese bestehen aus 5s, 16S und 23S RRNAs, während Eukaryoten auch 5,8- und 28S -Sorten haben. Biochemische Analysen haben einzigartige Eigenschaften von jedem ergeben:
5S rRNA – A 120 Nucleotid-RNA, die ribosomale Proteine bindet und in ein kompaktes 5-Helix-Bündel faltet, das in die zentrale Prognanzregion der 50er-Jahre-Untereinheit einpackt. Bietet ein Gerüst und hilft bei der Koordinierung von Intersubunit -Bewegungen.
16S rRNA – Bei ~ 1500 Nukleotiden, Es bildet den strukturellen Kern der 30S -Untereinheit. Enthält vier verschiedene Domänen, die sich in Sekundärstrukturelemente und Interdomänenbrücken falten, die für mRNA- und tRNA -Wechselwirkungen kritisch sind.
23S rRNA – Die größte rRNA bei ~ 2900 Nukleotiden. Innerhalb der 50er Jahre Untereinheit, Es verwendet eine charakteristische Gesamt -L -Form mit drei Domänen, die viele hochkonservierte Nukleotidsequenzen enthalten. Diese Nukleinsäuretaschen verwenden Konformationen, die zur katalysierenden Peptidbindungsbildung geeignet sind.
5.8S und 5S -rRNA – Wohnen in der 60er -Untereinheit. 5.8S rRNA nimmt eine Haarnadelhelix-Hairpin-Struktur an und hilft. Die 5S -rRNA, während kleiner, ist ähnlich strukturiert.
28S rRNA – Eukaryotischer Äquivalent von 23S -rRNA, die zum aktiven Zentrum beiträgt. Zusätzliche Expansion beherbergt zusätzliche Peptidtransfermaschinerie und regulatorische Elemente.
Strukturelle Kombination, Mutations- und Vernetzungsstudien haben genaue Orte jedes rRNA -Typs innerhalb der Untereinheiten gezeigt und veranschaulicht, wie ihre unterschiedlichen Topologien während der Dekodierung kooperieren, Korrekturlesen und Proteinproduktion.
Wie wird 16S -rRNA in der Mikrobiologie verwendet??
16S rRNA-Gen sequenzieren revolutionierte mikrobiologie, indem kultivierungsunabhängige mikrobielle Identifizierung und Klassifizierung ermöglicht werden. Regionen innerhalb des 16S -rRNA -Moleküls, die als hypervariable Regionen bezeichnet werden (V1-V9) Zeigen Sie die Variabilität der Nucleotidsequenz zwischen Organismen, sind jedoch in Arten hoch konserviert. Dies macht die V -Regionen ideale molekulare Marker.
Von PCR Verstärkung von 16S-rDNA aus einer Umweltprobe und Bestimmung der Abfolge der V3-V5-Regionen, Die erhaltene Signatur kann durch BLAST gegen Referenzdatenbanken charakterisierter mikrobieller Taxa ausgeführt werden. Die Datenbank, die mit der nächsten Übereinstimmung getroffen wurde.
Diese leistungsstarke Technik hat dazu beigetragen. Es ermöglichte eine schnelle Erkennung von Krankheitserregern aus klinischen und Lebensmittelproben ohne Kultivierung. Wissenschaftler verwendeten es, um zuvor versteckte Vielfalt in exotischen Umgebungen wie saurer Minenentwässerung oder hydrothermalen Lüftungsschlitzen zu untersuchen.
In jüngerer Zeit, Hochdurchsatz 16S-rRNA-Amplikon-Sequenzierung revolutionierte mikrobiome Forschung. Es ermöglicht die Profilierung ganzer mikrobieller Gemeinschaften direkt aus mikrobieller DNA in Proben ohne Verzerrung durch selektive Kultivierung. Die Ausgabe von Sequenzierungsplattformen der nächsten Generation wird mithilfe von Bioinformatik-Pipelines verarbeitet, um Sequenzen taxonomisch zuzuweisen und mikrobielle Assemblagen zwischen Umgebungen zu vergleichen, Wirtsnischen oder Behandlungsgruppen.
Zu den Einschränkungen gehören die Unfähigkeit, einige Arten und Primerverzerrungen zu lösen, die die Abdeckung beeinflussen. Andere Marker -Gene werden jetzt ebenfalls untersucht, Die 16S -RDNA -Analyse bleibt jedoch aufgrund der Datenbanktiefe der Goldstandard. Es stimuliert unzählige Entdeckungen fort, indem es in das dunkle Bereich der mikrobiellen Vielfalt auf der Erde hellt. Die Anwendungen reichen von der Diagnose über Bioprospektieren und Verständnis der Rollen von Mikroben in Ökosystemen, Gesundheit, und Krankheit.
Bietet rRNA Einblick in die Ursprünge des Lebens??
Einige Wissenschaftler spekulieren rRNA möglicherweise eine zentrale Rolle in den allerersten Lebensformen auf der Erde vor dem Auftreten von DNA und komplexen Zellen. Als selbstreplizierende und funktionell aktives Molekül, das die Grundlage der Proteinsynthese bildet, rRNA oder seine Vorfahren könnten ein Szenario darstellen, wie primitives Leben über Milliarden von Jahren Evolution zu einer größeren Komplexität gegangen ist.
Die laufenden Forschung zielt darauf ab, Hinweise aufzudecken, indem sie mehr über rRNA-Strukturfunktionsbeziehungen in verschiedenen Organismen, die heute leben.
Was sind fortgeschrittene Techniken zur Untersuchung von rRNA?
Angesichts der Komplexität von rRNA, Es werden verschiedene Methoden eingesetzt, um seine unzähligen Strukturfunktionsbeziehungen auf unterschiedlichen Maßstäben zu beleuchten:
- Die Sequenzierung der nächsten Generation bestimmt rRNA-Gensequenzen über Domänen hinweg, Unterstützung der Taxonomie und Charakterisierung von Varianten zwischen Arten.
- Kryo-Elektronenmikroskopie und Röntgenkristallographie ergeben nahezu atomare Auflösungsstrukturen ganzer Ribosomen oder Domänen, Aufschlussreiche rRNA-rRNA- und rRNA-Protein-Wechselwirkungen.
- Chemische Prüfkarten -Nukleotid -Beteiligung an der Faltung, Konformationsänderungen und Wechselwirkungen mit Liganden durch Reaktivitätsstellen gegenüber modifizierenden Wirkstoffen.
- Vernerzung in Verbindung mit Massenspektrometriespitzen rRNA -Regionen, die spezifische Proteine oder andere Biomoleküle in zusammengesetzten Partikeln oder Reaktionsintermediate kontaktieren.
- Einzelmolekül am Bund überwacht die Faltung einzelner rRNA-Moleküle und erkennt Konformationsverschiebungen bei biochemischen Prozessen wie der Translokation.
Das Integrieren von Erkenntnissen aus fortgeschrittenen Technologien auf Multi-Skala-Strukturfunktionsbeziehungen von RRNA entschlüsselt für die Translation und die zelluläre Homöostase mit beispiellose Tiefe wesentlich. Seine grundlegende Bedeutung inspiriert laufende Innovation.
Zusammenfassend, Während DNA als zentrales genetisches Lebensmaterial des Lebens am bekanntesten ist, Es ist der unbesungene Held – rRNA – Dies gewährleistet den kritischen Schritt, den DNA -Code in die funktionellen Moleküle zu verwandeln, die es Zellen und Organismen ermöglichen, durch seine wesentliche Rolle bei der Proteinproduktion über Ribosomen zu überleben. rRNA ist wirklich der unverzichtbare Motor des Lebens selbst.