- ส่วนประกอบของชุดรีเอเจนต์
| ข้อมูลจำเพาะ | 50ต | 100ต |
| แมว. เลขที่. | SN0229 | SN0230 |
| คอลัมน์สกัดดีเอ็นเอ (ชุด) | 50 (ชุด) | 100 (ชุด) |
| รีเอเจนต์บัฟเฟอร์ B | 20มล | 2×20 มล |
| รีเอเจนต์บัฟเฟอร์ C | 30 มล | 2×30 มล |
| ล้างบัฟเฟอร์ 1 | 15 มล | 2 × 15 มล |
| บัฟเฟอร์การชะล้าง | 20 มล | 20 มล |
| โปรตีนเค | 1มล | 1มล |
| ไลโซไซม์ | 1มล | 1มล |
| อาร์เนส เอ | 1มล | 1มล |
| คู่มือการใช้งาน | 1 | 1 |
- พื้นที่จัดเก็บ
ชุดรีเอเจนต์นี้ควรเก็บไว้ที่อุณหภูมิห้อง (15-25℃) และในสภาวะแห้ง, มีอายุการเก็บรักษาที่ 12 เดือน. คอลัมน์การทำให้บริสุทธิ์จากการสกัด DNA สามารถเก็บไว้ได้ 1 ปีในสภาพแวดล้อมที่เย็นและแห้ง. ไลโซไซม์, proteinase k และ rnase a มีสารกันบูด, จึงสามารถขนส่งได้ที่อุณหภูมิห้อง, แต่สำหรับการจัดเก็บระยะยาว, ควรเก็บไว้ที่ -20 ℃.
- คำแนะนำในการใช้ชุดรีเอเจนต์
3.1 ชุดรีเอเจนต์นี้มีไว้สำหรับการวิจัยอณูชีววิทยา และไม่ควรใช้สำหรับการวินิจฉัยหรือการรักษาโรค.
3.2 ส่วนประกอบบางอย่างในชุดรีเอเจนต์มีสารระคายเคือง. แนะนำให้ใช้มาตรการป้องกัน เช่น การสวมชุดป้องกันและแว่นตา.
3.3 ระหว่างการใช้งานชุดรีเอเจนต์นี้, เครื่องหมุนเหวี่ยงความเร็วสูง, อ่างอาบน้ำ (อาบน้ำโลหะ), เครื่องผสมน้ำวน, เอทานอลปราศจากน้ำ, น้ำปราศจากไอออนฆ่าเชื้อ, และผู้ใช้ต้องเตรียมท่อ EP.
- ข้อมูลเบื้องต้นเกี่ยวกับชุดรีเอเจนต์
ชุดนี้ให้วิธีการทำให้บริสุทธิ์อย่างรวดเร็วและมีประสิทธิภาพสำหรับการแยก DNA ออกจากของเหลวในร่างกายและของเหลววัฒนธรรมแบคทีเรียต่างๆ. มันใช้คอลัมน์การทำให้บริสุทธิ์จากซิลิคอนที่เลือกดูดซับกรดนิวคลีอิก. ด้วยโซลูชันบัฟเฟอร์เฉพาะ, ตัวอย่างดีเอ็นเอของแบคทีเรียสามารถสกัดได้ภายใน 30 นาที. กระบวนการทำให้บริสุทธิ์ทั้งหมดไม่จำเป็นต้องใช้รีเอเจนต์ที่เป็นพิษเช่นฟีนอลคลอโรฟอร์ม. DNA ที่สกัดสามารถใช้โดยตรงสำหรับการทดลองดาวน์สตรีมเช่น พีซีอาร์, การซับใต้, และคนอื่น ๆ.
- หลักและวิธีการทดลอง
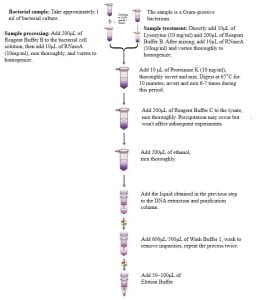
- กระบวนการสกัด
ก่อนเริ่มการทดลอง:
- สารรีเอเจนต์บัฟเฟอร์ B และ C อาจตกตะกอนภายใต้สภาวะอุณหภูมิต่ำ. เราขอแนะนำให้ร้อนที่ 65 ℃สำหรับ 5 นาที. หลังจากการตกตะกอนละลาย, สามารถใช้งานได้ตามปกติ.
- ก่อนใช้งาน, เพิ่มจำนวนเอทานอลที่ปราศจากน้ำ ล้างบัฟเฟอร์ 1ตามที่ระบุไว้บนฉลากขวด. ทำเครื่องหมายตรวจสอบฉลากเพื่อระบุการเพิ่มเอทานอลที่ปราศจากน้ำ.
- บัฟเฟอร์การชะล้างคือ0.1x โซลูชัน TEมี EDTA จำนวนน้อยที่สุด. หาก EDTA อาจส่งผลต่อการทดลองครั้งต่อไป, ขอแนะนำให้ทดแทนบัฟเฟอร์การกำจัดด้วยน้ำปราศจากไอออนที่ผ่านการฆ่าเชื้อ.
- การจัดการตัวอย่าง:
- ติด 1 ML ของวัฒนธรรมแบคทีเรีย, เครื่องหมุนเหวี่ยงที่ 12,000 รอบต่อนาทีสำหรับ 1 นาที, สำลักซุปเปอร์โนแลนต์ให้มากที่สุด, เพิ่ม 200μlของบัฟเฟอร์รีเอเจนต์ bไปยังสารละลายเซลล์แบคทีเรีย. โดยทั่วไป, RNA ที่เหลือมีผลกระทบน้อยที่สุดต่อการทดลองดาวน์สตรีม. หากจำเป็นต้องกำจัดสัญญาณรบกวน RNA, เพิ่ม 10μlของ rnasea (10มก./มล) ไปจนถึงส่วนผสม, ฟักตัวที่ 37 ° C สำหรับ 2 นาทีที่มีกระแสน้ำวนในช่วงเวลานั้น.
- หากตัวอย่างที่ถูกประมวลผลมีแบคทีเรียแกรมบวก, เพิ่ม 10μlของ lysozyme (10 มก./มล)และ 200μlของบัฟเฟอร์รีเอเจนต์ b. โดยทั่วไป, RNA ที่เหลือมีผลกระทบน้อยที่สุดต่อการทดลองดาวน์สตรีม. หากจำเป็นต้องกำจัดสัญญาณรบกวน RNA, เพิ่ม 10μlของ rnasea (10มก./มล) ไปจนถึงส่วนผสม, ฟักตัวที่ 37 ° C สำหรับ 15-30 นาทีที่มีกระแสน้ำวนในช่วงเวลานั้น.
- เพิ่ม 10μlของ proteinase k (10 มก./มล), คว่ำและผสมอย่างทั่วถึง, ย่อยที่ 65 ° C สำหรับ 2 นาที. ในช่วงเวลานี้, กลับตัวและผสมโซลูชันตัวอย่าง 6-7 ครั้งจนกระทั่งสารละลายตัวอย่างชัดเจนหลังจากการย่อยอาหาร.
- เพิ่ม 200μlของสารรีเอเจนต์บัฟเฟอร์ Cไปยังไลเซทและผสม. หากมีการตกตะกอนสีขาวปรากฏขึ้น, มันสามารถเหลือให้ชำระ; มันจะไม่ส่งผลกระทบต่อการทดลองที่ตามมาเมื่อการตกตะกอนหายไป.
- เพิ่ม 200μlของเอทานอล, ผสมให้เข้ากัน. การเร่งรัดบางอย่างอาจเกิดขึ้น แต่จะไม่ส่งผลกระทบต่อการทดลองในภายหลัง.
- ถ่ายโอนของเหลวที่ได้รับไปยังคอลัมน์การสกัดดีเอ็นเอ, ทิ้งไว้ที่อุณหภูมิห้องสำหรับ 2 นาที, เครื่องหมุนเหวี่ยงที่ 12,000 รอบต่อนาทีสำหรับ 30 วินาที. ทิ้งขยะที่เก็บรวบรวมและใส่เข้าไปในคอลเลกชันอีกครั้งลงในคอลัมน์การทำให้บริสุทธิ์สำหรับขั้นตอนต่อไป.
- เพิ่ม 600μl ของ Wash Buffer 1, เครื่องหมุนเหวี่ยงที่ 12,000 รอบต่อนาทีสำหรับ 30 วินาที, ทิ้งขยะ, และใส่คอลัมน์การสกัดดีเอ็นเอลงอีกครั้งลงในตัวยึด.
(บันทึก: ตรวจสอบให้แน่ใจว่ามีการเพิ่มเอทานอลลงในล้างบัฟเฟอร์ 1.)
- เพิ่ม 500μl ของ Wash Buffer 1 ไปยังคอลัมน์การสกัดดีเอ็นเอ, เครื่องหมุนเหวี่ยงที่ 12,000 รอบต่อนาทีสำหรับ 2 นาที, ขยายเวลาการหมุนเหวี่ยงตามความจำเป็นสำหรับเมมเบรนแห้ง.
- วางคอลัมน์การทำให้บริสุทธิ์สำหรับการสกัด DNA (เจ้าของ) ลงในหลอดหมุนเหวี่ยงใหม่, เปิด, และตั้งอุณหภูมิไว้ที่ 65°C เป็นเวลา 2 นาที. ขยายขั้นตอนนี้ตามความจำเป็นเพื่อระเหยเอทานอล, ป้องกันการตกค้างของเอทานอล.
- เพิ่ม 50-100μl ของบัฟเฟอร์การชะล้างไปยังเมมเบรนคอลัมน์, เครื่องหมุนเหวี่ยงที่ 12,000 รอบต่อนาทีสำหรับ 2 นาที.
(บันทึก: 1. การชะล้าง DNA ด้วย 50 μlของบัฟเฟอร์การชะ. 2. DNA ที่ออกจาก eluate สามารถนำไปใช้ใหม่ไปยังคอลัมน์การล้าง DNA Extraction, เครื่องหมุนเหวี่ยงอีกครั้งที่ 12,000 รอบต่อนาทีสำหรับ 2 นาทีเพื่อเพิ่มผลผลิตดีเอ็นเอ)








รีวิว
ยังไม่มีบทวิจารณ์