About the research
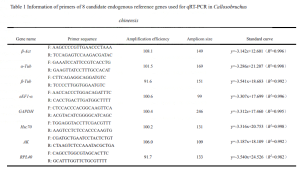
Currently used internal reference genes in qRT-PCR include various genes like ubiquitin-conjugating enzyme (UBC), 리보솜 RNA (rRNA), elongation factor 1-alpha (eEF1-α), ribosomal proteins (RPL), arginine kinase (AK), glyceraldehyde-3-phosphate dehydrogenase (갭DH), actin, and tubulin. Evaluating their stability is crucial because different species and conditions show variations in the stability of these reference genes.
The Callosobruchus genus includes pests like the Cowpea weevil, which damages seeds of leguminous crops such as mung beans and red beans. These weevils lay eggs on tender bean pods in fields, and when the larvae hatch, they tunnel into the beans. Eggs and larvae can also enter storage after harvesting, causing a second wave of infestation, which can be more severe if not controlled promptly. These pests are adept at hiding, have a wide distribution, and possess a strong ability to cause repeated infestations, significantly impacting the yield and quality of leguminous crops like mung beans and red beans. Although there are studies on the gene expression of these weevils, the exploration of internal reference genes remains incomplete. This study identifies eight candidate internal reference genes based on previous laboratory research: α-tubulin, β-tubulin, β-actin, elongation factor (eEF1-α), arginine kinase (AK), heat shock protein 70 (Hsc70), glyceraldehyde-3-phosphate dehydrogenase (갭DH), and ribosomal protein 40 (RPL40). These genes were assessed for their expression levels using qRT-PCR. Through GeNorm, Normfinder, 베스트 키퍼, and the online tool RefFinder, along with ΔCt values, the study explores the expression stability of these candidate internal reference genes in different developmental stages of the Cowpea weevil and in various tissues of adult weevils. This research aims to provide reliable internal reference genes for future studies on gene expression and function in the Cowpea weevil.
About the test
Total RNA was extracted from SENO animal tissues using a kit.
Different tissues from Cowpea weevils at various developmental stages and adult weevil tissues were used to synthesize cDNA using the HiScript® III 1st Strand cDNA Synthesis 전부 (+gDNA wiper).
This process was repeated three times following the kit instructions. The concentration and quality of the extracted total RNA were checked using a spectrophotometer and agarose gel electrophoresis.
Eight protein-coding genes (β-Act, β-Tub, α-Tub, AK, 갭DH, RPL40, Hsc70, eEF1-α) were chosen as candidate genes.
Quantitative primers were designed using Beacon Designer 7 software and synthesized by a biotech company.
The cDNA templates were diluted in a 1:10 gradient to create five concentrations for qRT-PCR reactions, constructing standard curves, and calculating primer amplification efficiency E.
The qRT-PCR results were used to plot standard curves for the primers, and the amplification efficiency E was calculated using the slope of the standard curve (E = (10^-1/slope^-1) × 100).
The qRT-PCR reaction mixture (20 μL) comprised ChamQ Universal SYBR qPCR Master Mix (10 μL), ddH2O (8.2 μL), PCR Forward Primer (0.4 μL, 10 μmol/L), PCR Reverse Primer (0.4 μL, 10 μmol/L), and cDNA (1 μL). The reaction conditions were: initial denaturation at 95℃ for 30 에스; 40 cycles of denaturation at 95℃ for 10 에스, annealing/extension at 60℃ for 30 에스; and a melting curve from 60 to 95℃ for 30 에스. Amplification and melting curves for qRT-PCR were confirmed. Each sample underwent three technical replicates.
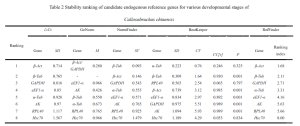
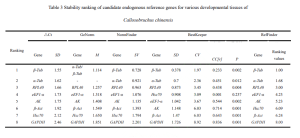
The data from qRT-PCR Ct values were analyzed using △Ct, 표준화 (Vandesompele et al., 2002), 베스트 키퍼 (Pfaffl et al., 2004), and NormFinder (Andersen et al., 2004). △Ct and BestKeeper used raw Ct values, while GeNorm and NormFinder transformed the data into 2^-△Ct. △Ct calculated the standard deviation (SD) of Ct value differences between each candidate reference gene pair for stability ranking—smaller SD indicating more stable genes. GeNorm ranked candidate reference gene stability using stability value (중) 계산. When M < 1.5, the gene was considered suitable for reference gene selection, with lower M values indicating higher stability. 게다가, GeNorm determined the optimal number of reference genes using the pairwise variation value V. If the Vn/n+1 value was below the threshold of 0.15, n was deemed the optimal number of reference genes, without the need for adding the n+1th reference gene for correction. BestKeeper software evaluated the stability of candidate reference genes based on Ct value standard deviation (SD), coefficient of variance (CV), correlation coefficient (CC[아르 자형]), and significance level (피). Genes with SD < 1 were considered stable, with smaller SD and CV values indicating higher stability. A higher CC[아르 자형] value, under P < 0.05, signified a greater correlation with other genes, making it a good candidate as a control gene. 마지막으로, RefFinder, an online tool, comprehensively analyzed reference gene stability, providing a reliable composite ranking index—lower values indicated more stable internal reference genes.
The melting curve analysis from qRT-PCR confirmed that the eight candidate internal reference genes (β-Act, β-Tub, α-Tub, AK, 갭DH, RPL40, Hsc70, eEF1-α) had no non-specific amplification, demonstrating good specificity for subsequent experiments (Figure 1). The results from standard curves for the eight candidate internal reference genes showed a good linear relationship within the dilution gradient, meeting the amplification conditions for qRT-PCR.
결론
현재, 유전자 발현 연구는 주로 QRT-PCR 기술에 의존합니다, commonly using reference genes to correct errors introduced during sample detection. 초기 연구는 일반적으로 세포 활성을 유지하는 데 필수적인 하우스 키핑 유전자가 다른 조건 하에서 안정적인 발현 패턴을 나타냅니다., 보편적 인 기준 유전자로 간주됩니다.
하지만, 고 처리량 시퀀싱의 발전으로, 더 많은 연구는 하우스 키핑 유전자가 항상 안정적이지 않다고 제안합니다.. 최적의 기준 유전자는 종종 다른 조건에서 다른 종마다 크게 다릅니다..
표적 유전자의 발현을 연구 할 때, 검증없이 이전에 게시 된 참조 유전자를 사용하면 편향된 결과가 발생할 수 있습니다.. 따라서, it’s essential to reselect reference genes.
Cowpea Weevil의 경우, it was found that the pairwise variation value Vn/n+1 in the GeNorm software’s pairwise difference analysis was consistently less than the default value of 0.15 다른 발달 단계와 성인 바구미의 다양한 조직에서. 그러므로, 데이터 보정을위한 세 번째 기준 유전자를 포함 할 필요가 없습니다.; 가장 적합한 수의 기준 유전자는 2입니다. CT를 통한 포괄적 인 평가, 표준화, Normfinder, 베스트 키퍼, 그리고 Reffinder Online 도구는 Cowpea Weevil의 다른 발달 단계에서 유전자 발현 연구를위한 내부 기준 유전자로서 β-act 및 β-tub를 사용하는 것을 제안합니다.. 비슷하게, 성인 바구미의 다양한 조직의 경우, 내부 참조 유전자로서 β- 튜브 및 α- 튜브를 권장 하였다.
RPL40은 성인 cowpea 바구미의 다른 조직에서 내부 기준 유전자 중 하나로 사용하기에 비교적 적합한 것으로 평가되었습니다.. 하지만, 그것은 바구미의 다른 발달 단계 중에서 가장 안정적인 유전자 중 하나 인 것으로 보인다..
반면에, GAPDH는 성인 cowpea eevils의 상이한 조직들 사이에서 발현에서 가장 적은 안정성을 보여 주었고, 바구미의 다른 발달 단계에서 유전자 발현 연구에 대한 기준 유전자로서 부적합했다.. Colaphellus Bowringi 및 Sympiezomias velatus와 같은 다른 콜로오 테라 곤충에서도 비슷한 결과가 관찰되었습니다. (So et al., 2015; Li et al., 2018).
이 연구는 QRT-PCR을 사용하여 다른 발달 단계 및 COWPEA 바구미의 다양한 조직에서 유전자 발현 수준을 정확하게 분석하기위한 정확하고 신뢰할 수있는 내부 기준 유전자를 제공합니다.. 하지만, 이러한 결과가 다른 실험 조건에 적용되는지 여부는 추가 조사가 필요합니다..
Cowpea Weevil에서 내부 기준 유전자의 발현은 다른 실험 조건에서 크게 다릅니다.. 이 연구는 5 가지 방법을 활용하여 다른 발달 단계에서 8 개의 후보 내부 참조 유전자의 발현 안정성을 평가하고 Cowpea Weevil의 다양한 조직. 결과는 Genorm 소프트웨어가 다른 조건에서 V2/3 유전자 쌍별 차이 값을 계산했음을 시사합니다., 이 모든 것은 임계 값보다 낮았습니다 0.15. 그러므로, 상대 유전자 발현을 연구하기위한 최적의 기준 유전자는 2입니다.. 구체적으로, Cowpea Weevil의 다른 발달 단계, 내부 참조 유전자로 β-Act 및 β-tub를 사용하는 것이 좋습니다.. 그 동안에, 성인 바구미의 다양한 조직의 경우, β- 튜브 및 α- 튜브는 내부 참조 유전자로 권장됩니다.
