クラミジア検出のための qPCR 手順には、2 つの主要なステップが含まれます: DNA 抽出とそれに続く DNA の増幅と検査. 最初は, DNA 抽出に関する学習コンテンツの共有に重点を置きます。. この記事では、主に理論的基礎と使用される試薬の紹介について詳しく説明します。.
目次
トグルDNA 抽出の理論的精製原理 実験を行う前に, 基本原理と運用手順を理解することは有益です. 実験を理解するのに役立ちます, 潜在的な結果を予測する, 単に反復的な演算子になることを避けます.
DNA抽出の基本原理
DNA構造の完全性: 例えば, qPCR プローブベースのクラミジア検出の背後にある原理には、クラミジア ゲノム内の 16SrRNA コード領域を増幅することによるクラミジアの特異的検出が含まれます。. クラミジアの特異的増幅は520nmで検出可能 (FAMチャンネル). 不完全なクラミジア DNA は、特徴的な増幅と検出を受けることができません。.
考慮事項:
(1) これを確実にするために, 私たちの実験手順では、DNA の分解を防ぐために穏やかな取り扱いが必要です.
(2) DNase の不活化に注意が必要.
(3) 対応する抽出試薬キットを含まずに qPCR クラミジア試薬キットのみを購入した場合, テストキットの適用性をテストするだけでなく、抽出プロセスの有効性を検証する必要もあります.
1.1.2 DNAの純度: DNA を妨害する可能性のある他の大きな分子物質を排除するよう努めます。, したがって、その後の実験への影響を回避できます. 抽出されたサンプルには、酵素を阻害する有機溶媒や高濃度の金属イオンが含まれていないことを確認してください。.
考慮事項:
(1) 細胞サンプル内の総細胞数を制限する場合にも同様の考慮事項が適用されます。, 他の大きな分子物質内の非標的 DNA が実験に影響を与える可能性があるため.
(2) 品質管理上の注意事項 (品質管理): サンプリングは過剰であってはなりません. QC の代表性を高めるためにテスト量を増やすことが望ましい場合もありますが、, このような場合には注意が必要です.
(3) サンプルを理解することが重要です; 酵素を阻害する有機溶媒や金属イオンが含まれている場合, これらの物質が実験前に効果的に除去できるか、細胞培養法によって濃度を低減できるかどうかを確認する必要があります。.
(4) 他の核酸からの汚染は一般的な干渉因子です.
特に, クラミジアまたはクラミジア陽性の検体をボルテックスして取り扱うとき, エアロゾルが発生する可能性がある. したがって、, クラミジア検査の際に, バイオセーフティキャビネット内での厳格な無菌操作が重要です, ボルテックス後は素早く遠心分離することをお勧めします.
DNA 抽出手順
DNA 抽出はわずか 2 ステップで完了します: 細胞溶解と精製.
- 細胞溶解 細胞溶解は、サンプル細胞の構造を破壊します。, DNA およびその他の細胞成分を溶液中に放出する. DNA 溶解の方法には機械的方法が含まれます。, 化学薬品, および酵素プロセス.
- 機械的方法では液体窒素による粉砕が行われることが多い.
- 化学的方法のような CTAB そして SDS 細胞膜を溶解することを目的としています. CTABはカチオン性洗浄剤として機能します, SDSは陰イオン洗浄剤です, どちらも細胞膜の溶解を助けます. クレンザーはタンパク質を変性させる, 膜構造を破壊し、核酸に結合したタンパク質を放出する.
- 酵素作用, プロテイナーゼKによる消化など.
考慮事項:
(1) 当社の細胞治療製品の検出ターゲットについて, 特に細胞上清, 酵素消化はより穏やかで簡単なアプローチです.
(2) 酵素活性は温度や塩イオンなどの環境要因に影響されます。. 有機溶媒や高濃度の塩イオンによる潜在的な影響を回避するには、最初のサンプルを理解することが重要です. 適切なバックグラウンド環境を作成するためにバッファを追加できます。.
DNA 精製 精製は、タンパク質などの他の細胞成分から DNA を分離します。, 脂質, 多糖類, およびRNA.
細胞構造が溶解すると, DNA を単離する基本的なアプローチは、DNA と RNA などの他の成分の間の物理的および化学的特性の違いを利用することに依存しています。, タンパク質, そして脂質.
- DNA は 0.14mol/L NaCl 溶液中で最も溶解度が低くなります。. DNAを高塩濃度溶液に溶解すると、高塩に不溶な不純物を除去できます。. 低塩溶液を使用して DNA を沈殿させると、低塩に可溶な不純物が除去されます。.
- ほとんどのタンパク質は60~80℃の温度に耐えられません。, 一方、DNAは80℃を超えると変性します。.
- DNAはアルコールに溶けない, しかし、細胞内の特定のタンパク質はその中に溶解する可能性があります.
考慮事項:
(1) これらのポイントを理解することで、さまざまな精製方法を理解することができます。.
(2) 抽出・精製の有効性を検証する場合, 実験原理に基づいて、特定のステップの変更または追加が必要になる場合があります。.
クラミジア DNA 検査のための一般的な DNA 抽出方法, クラミジア検査キットメーカーが提供する主流の検査方法を理解することが重要です. 一般的な方法には、ビーズベースの方法と沈殿ベースの方法が含まれます。. ろ過方法, コストが高いため, 現在は考慮されていません, コストを考えながら, 遠心カラム法は個人的な好みによるかもしれません.
沈殿法 沈殿法には、タンパク質を除去するためのフェノール/クロロホルム抽出と DNA を沈殿させるためのエタノール/イソプロパノールが含まれます。. フェノールは水相から変性タンパク質を抽出します, DNaseによるDNA分解の阻害. クロロホルムは有機相と水相の分離を促進します, 残留フェノールの除去.
考慮事項:
(1) クロロホルムが怖いので、換気フードの外では不安になります.
(2) 大量のサンプルを処理できる能力を考慮する, この方法を試してみたいと思っています.
(3) 沈殿法だと不純物が析出してしまわないか少し不安, したがってさらなる研究が必要です.
遠心カラム法
この方法には、細胞構造を迅速に破壊し、細胞ヌクレアーゼを不活性化する独自の結合溶液/プロテイナーゼ K が含まれます。. ゲノム DNA は、高塩分状態の遠心カラム内のシリコンベースの膜に選択的に付着します。. 一連の迅速な洗浄と遠心分離のステップを経て, 阻害物質は細胞の代謝物やタンパク質とともに除去されます, 精製されたゲノム DNA を残す, その後、低塩溶出バッファーを使用してシリコンベースのメンブレンから溶出されます。.
考慮事項:
(1) 非常に安全です, ただしサンプル量は少し少ないかもしれません.
(2) 採取管の容積が限られているため、操作スペースが制限される.
(3) 確実に検出するには, DNAを濃縮する必要がある, これにより、ピペッティング量がさらに減少します. ピペットを使いこなすことが重要です.
(4) コストを考慮して, 遠心カラム法は一時的な選択です.
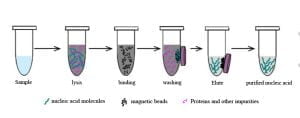
磁気ビーズ法
磁気ビーズ法の主なステップには溶解が含まれます。, バインディング, 洗浄, そして溶出. 具体的な手順についてはここでは詳しく説明しません.

このメソッドで使用される溶解バッファーはタンパク質を変性します。, 細胞溶解と DNA に結合したタンパク質の変性を引き起こす. 磁性ビーズがDNAを特異的に吸着します. 洗濯を通して, RNAやタンパク質などの不純物, DNAとは別に, 削除されます. それから, 溶出液はビーズに結合した DNA を解離します。, その結果、高純度で濃縮された DNA が得られます。 PCR 増幅.
ナノテクノロジーとバイオテクノロジーを組み合わせた磁気ビーズ法, 常磁性を示す生物学的に活性な物質を主成分とする磁性ナノ粒子を使用する. これらのビーズには、陽イオン交換が可能な表面層があります。, 核酸の吸着を促進する. 表面素材は水素結合と静電相互作用を通じて核酸と結合します。. 精製原理には、高塩分条件で核酸を結合し、低塩分環境で核酸を溶出することが含まれます。. さらに, 磁性ビーズは磁場条件下で凝集または分散します, 遠心分離などの手動操作が不要になります。.
考慮事項:
(1) 磁気ビーズ法の核心はビーズにあります. 一部のキットには限られた数のビーズが含まれています, 専門家に独自のシステムを確立するよう促す. 彼らの活躍を楽しみにしています.
(2) マグネットスタンドを選ぶときは, 目に見えるものを選ぶ.
DNA純度評価
DNA抽出後, 純度の評価, 集中, そして誠実さが不可欠です. 一般的な方法には次のものがあります。:
- ゲル電気泳動: この方法ではアガロースゲルを使用して残留 RNA を測定します。, タンパク質, フェノール, 抽出された DNA 内の他の代謝物. さらに, バンドの明るさは DNA の完全性を示します. ゲル電気泳動, バンドの明るさをDNAマーカーと比較することにより, DNA濃度を大まかに推定します.
- 分光測光法: DNA または RNA 塩基のベンゼン環は 260nm の紫外領域を強く吸収します。. タンパク質は280nmで最大吸収を示します, 一方、塩と小分子は 230nm に集中します. 分光光度計の使用, 原理は吸光度が DNA 濃度と相関するということです. DNA 純度の評価には通常、OD260 の測定が含まれます。, OD280, およびOD230値, 比率に基づいて純度を判断する.
いつもの, 純粋な DNA の OD260/OD280 比は約 1.8. 上記の比率 1.9 DNA 分解または RNA 汚染を示唆する, 下にいる間 1.7 タンパク質の残骸を示します. ゲル電気泳動は、純度と完全性を評価するために最初に実行されることがよくあります。, その後、UV分光光度法で濃度を測定します.
QBITの検出方法
この技術は、特定の標的分子に結合すると蛍光を発する蛍光色素を利用します。. 蛍光値を測定し、濃度データに変換する装置です。. 分光測光法との比較, この方法はより感度が高いです, 特定の, 正確な検出.
考慮事項:
(1) コストを考慮して, メソッド 1.5.1 そして 1.5.2 現在、抽出方法の開発中により便利になっています.
(2) 抽出効果を確認するには, その後の品質管理措置が必要である. 独自にメソッドを開発する場合, 効果的な品質管理を選択することが不可欠です.
DNA 抽出および精製試薬情報の紹介
理解を深めるために使用できるさまざまな試薬の概要をまとめました. このまとめは参考用です, オリジナルではない, 教育交流のみを目的としたもの.
プロテイナーゼ K プロテイナーゼ K, 白色菌から分離, 高い比活性を持つ強力なタンパク質消化酵素です, DNA抽出に不可欠な. 広いpH範囲内で活性を維持します (4-12.5) そして高温で (50-70 摂氏). EDTA などのキレート剤や SDS などの脱イオン剤では不活性化されません。.
プロテイナーゼK, 広い切断活性を持つセリンプロテアーゼ, 脂肪族および芳香族アミノ酸のカルボキシル末端のペプチド結合を切断します。. 細胞膜や核膜上のさまざまな膜タンパク質や糖タンパク質を消化し、DNA に結合したヒストンも消化します。.
塩化ナトリウム (塩化ナトリウム) NaClはDNAに結合したタンパク質の除去を助けます. DNAのマイナス電荷を中和することで, 分子の凝集を可能にし、タンパク質を水層に溶解した状態に保ちます。, アルコール中のDNAとの沈殿を防ぐ.
細胞が低張または高張の溶液にさらされる場合、塩濃度は重要な役割を果たします。, 浸透につながる.
EDTA EDTA は、Mg2+ や Ca2+ などの二価陽イオンをキレートします。, 酵素の中に存在するもの, DNase および RNase の酵素活性を低下させる. 例えば, DNase 酵素は、その活性の補因子として Mg2+ イオンを必要とします. EDTA による Mg2+ イオンのキレート化により DNase が不活性になります, DNAを保護する. マグネシウムイオンは核酸とタンパク質の凝集にも必要です, 一方、カルシウムイオンは細胞壁の完全性と膜の安定性に不可欠です.
フェノール, 水に溶けない有機溶媒, DNA精製にはクロロホルムおよびイソアミルアルコールとともに使用されます, タンパク質と多糖類の汚染物質を除去する. 細胞抽出物と一緒に振った場合, 細胞の非極性成分はフェノール中で分別されます, 極性成分を水中に残す. フェノールは非極性溶媒であるため、DNA はフェノールに溶けずに残ります。. 一方で, タンパク質は、その長鎖に異なるアミノ酸があるため、極性基と非極性基の両方を含みます。. タンパク質の二次構造への折り畳み, 三次, そして四次構造はアミノ酸の極性にも依存します.
フェノール混合物による遠心分離: とクロロホルム:
イソアミルアルコールの 25:24:1 比率により 3 つの層が生成されます: 水性, 間期, そしてオーガニック. DNA, 負に帯電し、中性からアルカリ性の pH で極性がある, 親水性を保ち、水相に保持されます。. タンパク質中の疎水性アミノ酸が水溶液中のDNAを保護する. しかし, 変性時, 非極性核酸が露出している, タンパク質と特定の多糖類が中間相で沈殿する原因となります。.
フェノールとクロロホルムの組み合わせにより、ポリフェノールの分布が減少します。(あ) 有機相のRNAとmRNAを分離し、界面での不溶性RNA-タンパク質複合体の形成を最小限に抑えます。. フェノールは水相の約 10 ~ 15% を保持します。, 同様のRNA損失を引き起こす. クロロホルムは水分の滞留を防ぎます, それにより収量が向上します.
酸性フェノールはDNAを溶解するため、中性フェノールのみを使用してください。, またはフェノール, 酸化作用を介して, キノン類に変化する, 核酸を分解するフリーラジカルを生成する.
クロロホルム
クロロホルムは無極性です (疎水性の) 非極性タンパク質と脂質が溶解する溶媒. これにより、脂質と細胞破片の有機相への分布が促進されます。, 水相で分離されたDNAを保護する. 比較的高密度で (1.47 g/cm3), クロロホルムは 2 つの液体を確実に分離します, 有機相と水相を明確に分離できるようにし、有機相との相互汚染を最小限に抑えながら水相の除去を支援します。. クロロホルムは本質的に揮発性であるため、, 後続のプロセスを妨げません.
補足: DNA 抽出にフェノールとクロロホルムを使用してタンパク質を除去する方法?
フェノールとクロロホルムは非極性分子です, 一方、水は極性分子です. タンパク質と水の溶液がフェノールまたはクロロホルムと混合する場合, タンパク質分子間の水分子がフェノールまたはクロロホルムによって置換される, タンパク質が水和状態を失い、変性を引き起こす. 遠心分離後, 変性タンパク質, 水よりも密度が高い, 水相から分離し、その下に沈殿する, 水相に溶解したDNAからの分離. フェノールとクロロホルム, 有機溶剤であること, タンパク質の除去には利点と欠点があります. フェノールはより顕著な変性効果を持っていますが、部分的に水相に溶解します。, 約の損失が発生する 10% に 15% 水相中のDNAの. クロロホルムの変性効果はフェノールほど効果的ではありません, でも水には溶けない, したがってDNAを持たない. したがって, フェノールとクロロホルムを組み合わせると、抽出プロセス中に最良の結果が得られます。.
イソペンタノール (イソプロパノール)
クロロホルムが空気にさらされると有害なガスが発生する, ホスゲン. クロロホルムのみを使用する場合, ガスが閉じ込められると泡立ちや泡立ちが発生します, 2 つのフェーズ間で DNA を正確に精製することが困難になる. この時点で, クロロホルムとイソプロパノールまたはオクタノールを組み合わせると、抽出プロセス中の溶液の乳化が防止されます。.
RNase A
RNase A 3 つの加水分解を触媒するエンドリボヌクレアーゼです。′,5′-RNAの5番目のホスホジエステル結合′-二段階反応でのエステル結合. 最初のステップはリン酸転移です, ピリミジン 2 で終わるオリゴヌクレオチドの終結をもたらす′,3′-環状リン酸. 第 2 ステップは、環状リン酸を加水分解して末端 3-ホスホジエステルを生成することです。. DNAには重要な2が欠けている′-おお, RNase Aによる消化に耐性を与える.
イソプロパノール/エタノール
エタノールは抽出溶液から DNA を沈殿させます. DNA, ホスホジエステル骨格を持つ, 本質的に親水性です. 水分子は水素結合により DNA の周囲に水和シェルを形成します. DNAを沈殿させるためにイソプロパノール/エタノールを使用します, 水和シェルを破壊する. イソプロパノールは DNA 沈殿に適しています. より少ない体積が必要です (0.6–0.7 倍の上清の体積). RNA, 追加の 2’OH, 水に溶けやすく、選択的に沈殿する傾向があります。, DNAを残す. イソプロパノールはクロロホルムなどの非極性溶媒を溶解します, これにより、前のステップからの不純物が除去されます。.
通常, 冷イソプロパノールが使用される, しかし研究者らは、多糖類の沈殿を防ぐために室温を推奨しています。. 低温では DNA 収量が増加しますが、, 不純物が増える可能性があります.
イソプロパノール (利点: 必要な量が少なくて済む, 急速な降水, 低濃度に適しています, 大量の DNA サンプル; 短所: DNAと塩が共沈しやすい; 複数回の洗濯が必要 70% エタノール) エタノール (利点: 塩による最小限の沈殿, DNA沈殿中の残留エタノールは容易に蒸発します, その後の実験には影響しません; 短所: 全体のボリュームが大きくなる, 効果的な沈殿を得るには、-20°Cでの長期保管が必要です, 必要 70% エタノール洗浄)
酢酸ナトリウム/酢酸アンモニウム/酢酸カリウム/塩化ナトリウム/塩化リチウム/塩化カリウム
抽出手順における塩は、DNA 糖リン酸骨格の電荷を中和することによって機能します。. pHでの酢酸ナトリウム 5.2 核酸の沈殿にはエタノールと併用されるのが一般的です. 溶液中, 酢酸ナトリウムはNa+に解離し、 [CH3COO]- イオン. 正に帯電したナトリウムイオンは、PO3 の負電荷を中和します。- 核酸の糖リン酸骨格の, DNA分子間の反発を減らす. これにより、DNA分子の親水性が大幅に低下します。, したがって、水への溶解度が大幅に低下します.
エタノール
DNA 沈殿物を洗浄します。 70% 余分な塩分を除去するためのエタノール, 遠心, そしてエタノールを捨てます, 沈殿物中に DNA を残す. 沈殿物を自然乾燥または真空乾燥します。. 過度の乾燥は、後で DNA を溶解するのが困難になる可能性があるため避けてください。.
トリス-EDTA (ザ) 緩衝液/滅菌水
以前の DNA 単離法では, DNAは乾燥させて保存する必要がある, その後、必要に応じて希釈します. 最近では, 長期保管用, DNA は、pH を維持し分解を防ぐ緩衝液に保存することが賢明です。. TE バッファーには Tris が含まれています (10 mM) とedta (1 mM), ここで、Tris は緩衝剤として機能し、EDTA はキレート剤として機能します。. DNA分離用, 通常、pH は次のように設定されます。 7.5-8.5. TE バッファーの弱アルカリ性は、酸加水分解の可能性を防ぐのに役立ちます。, 水中でのDNAのさらなる安定化.
TE バッファーのトリスアミノ成分は、固体および液体溶液中の放射線損傷から DNA 鎖を保護します。. 放射線がフリーラジカルを生成すると, DNA鎖を傷つける可能性があります. したがって, 室温の液体溶液中, トリスはヒドロキシルフリーラジカルを除去することによって作用します.
EDTA は、DNase または RNase 活性に必要な溶液中の Mg2+ イオンをキレート化することを目的としています。, DNase または RNase 攻撃から DNA を保護する.
滅菌水は短期間の DNA 保存に使用可能. DNA の保存に TE バッファーを使用する場合, EDTA 濃度を下げるために、滅菌水でさらに希釈する必要があります。. これにより、PCR 中にマグネシウムイオンがポリメラーゼ活性に利用できるようになります。. 後で DNA の配列を決定する必要がある場合、TE のバッファー成分が DNA の増幅を妨げる可能性があります。.