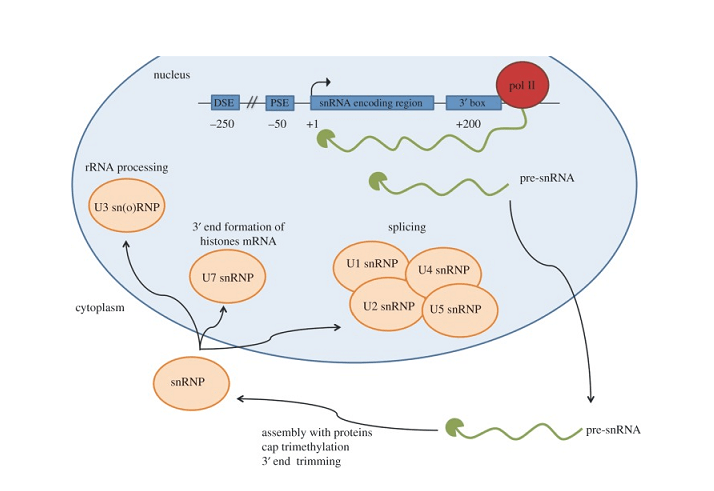
Singoli RNA nucleari, o snRNA, svolgono un ruolo cruciale all'interno delle cellule nonostante la loro breve lunghezza. Come molecole di RNA non codificanti presenti esclusivamente nel nucleo, Gli snRNA partecipano a modifiche post-trascrizionali essenziali che aiutano a sintetizzare le proteine funzionali. Ci sono due classi principali di snRNA – U1, U2, U4, U5, e u6 che formano le piccole ribonucleoproteine nucleari (snrnps) coinvolto nella giunzione, e gli snornas H/ACA e C/D Box che guidano le modifiche chimiche di altri RNA.
Cos'è il sequenziamento dell'RNA a nucleo singolo (snrna-seq)?
Sequenziamento dell'RNA a nucleo singolo, o snrna-seq, è una tecnica innovativa che analizza il trascrittoma nucleare isolato dalle singole cellule. Questo approccio aiuta a ridurre al minimo le risposte allo stress trascrizionale che possono distorcere i risultati durante la dissociazione. Rispetto al tradizionale sequenziamento dell'RNA a cella singola (scrna-seq), SnRNA-seq mostra alcuni benefici che migliorano la sua utilità per lo studio dell'eterogeneità cellulare.
Qual è la differenza tra snrna-seq e scrna-seq?
Sequenziamento dell'RNA a nucleo singolo (snrna-seq), noto anche come snrna-seq o snuc-seq, è un metodo di sequenziamento dell'RNA innovativo utilizzato per profilare l'espressione genica nelle cellule che sono difficili da isolare. Questa tecnica funge da potente alternativa al sequenziamento dell'RNA a cella singola (scrna-seq) Analizzando i nuclei anziché le cellule intatte. Ma cosa distingue esattamente snrna-seq da scrna-seq? Approfondiamo le differenze chiave.
ScrNA-seq misura trascrizioni sia citoplasmatiche che nucleari, Fornire una visione completa del profilo di espressione genica di una cellula. Al contrario, SnRNA-seq misura principalmente le trascrizioni nucleari, mentre si concentra sull'analisi dell'RNA all'interno del nucleo stesso. Questo approccio mirato minimizza il verificarsi dell'espressione genica spuria, Mentre i ribosomi maturi risiedono nel citoplasma. Perciò, Eventuali fattori di trascrizione espressi dopo il processo di dissociazione non possono essere tradotti, impedire la trascrizione dei loro obiettivi a valle.
In che modo SnRNA-seq ospita tipi di tessuto impegnativi?
Per uno, SnRNA-seq ospita una più ampia varietà di tipi di tessuto che può essere difficile dissociarsi in singole cellule. Tessuti complessi con dense matrici extracellulari come il cervello, rene, e il cuore può essere analizzato in modo più efficace usando snRNA-seq a causa del suo protocollo di dissociazione più mite. Ciò preserva l'integrità di tipi di cellule sensibili che possono sottoporsi a cambiamenti trascrizionali o degradarsi durante la dissociazione più dura richiesta per ScrNA-Seq.
Quali sono i vantaggi dell'utilizzo di snrna-seq per campioni archiviati?
SnRNA-seq supporta anche lo studio di campioni clinici archiviati. I tessuti congelati o fissi che sarebbero incompatibili con scrNA-seq possono essere profilati finché i nuclei intatti possono essere isolati. Ciò espande l'ambito di possibili applicazioni di ricerca rispetto ad altri metodi di sequenziamento che richiedono singole cellule appena isolate. In particolare, SnRNA-seq ha permesso agli investigatori di analizzare il tessuto cerebrale umano post mortem per scoprire la diversità neuronale a livello di singola cellula.
Quale prospettiva unica fornisce snrna-seq?
Piuttosto che una visione completa dell'intero trascrittoma, SnRNA-seq fornisce approfondimenti focalizzati su processi nucleari e interazioni modulate da SnRNA. Offre una prospettiva unicamente ingrandita sui meccanismi regolatori all'interno dello spazio nucleare confinato. L'applicazione di snrna-seq a campioni di tessuto complessi ha rivelato nuovi tipi di cellule e stati che potrebbero non essere rilevati usando lo scrna-seq da solo. Tali scoperte approfondiscono la nostra comprensione dell'organizzazione cellulare e della specializzazione, espandere le frontiere della medicina di precisione.
Come può snRNA-seq aiutare l'espressione genica del profilo in campioni stimolanti?
Le capacità di profilazione nucleare di snRNA-seq si dimostrano particolarmente preziose per i campioni che sono problematici da analizzare utilizzando approcci convenzionali scrNA-seq. Alcuni tipi di tessuto pongono difficoltà tecniche a causa di qualità come la composizione della matrice densa, fragilità alla dissociazione, o incompatibilità con i trattamenti enzimatici richiesti per l'isolamento a cellula singola. I campioni archiviati conservati attraverso la fissazione presentano anche sfide.
Come è stato usato SnRNA-seq per studiare il tessuto cardiaco dei mammiferi?
Il tessuto cardiaco dei mammiferi esemplifica un sistema intrattabile per lo scrNA-seq ma suscettibile all'analisi snRNA-seq. Le cellule muscolari cardiache formano un'intricata rete interconnessa non facilmente interrotta senza danno cellulare. Tuttavia, In 2020 I ricercatori tedeschi hanno sequenziato con successo il primo cuore dei mammiferi adulti usando snrna-seq. Isolando i nuclei anziché i cardiomiociti interi, Hanno ottenuto distribuzioni pratiche di popolazioni cellulari distintive all'interno del miocardio. Tale profilazione su larga scala sarebbe poco pratico attraverso la metodologia scrna-seq.
In che modo snRNA-seq a beneficio dello studio delle popolazioni neuronali?
Le popolazioni neuronali rappresentano un'altra classe che beneficia delle capacità di snrna-seq. Il cervello è costituito da neuroni integralmente cablati non prontamente dissociati come singole cellule intatte. Il tentativo di farlo può indurre le risposte allo stress che si distinguono. Tuttavia, studiare l'eterogeneità trascrittomica dei sottotipi neurali informa la nostra comprensione della neurobiologia, meccanismi di malattia, e altro ancora. Protocolli SnRNA-seq ottimizzati per i campioni cerebrali umani archiviati e appena resecati appena resecati con una risoluzione a cella singola.
In che modo snRNA-seq consente l'analisi di campioni clinici congelati?
Allo stesso modo i campioni clinici congelati pongono i limiti superati da snrna-seq. Gli studi sulla malattia del fegato che confrontano i nuclei polmonari sani e fibrotici rilevati tramite snRNA-seq hanno rivelato l'identificazione imparziale dei tipi di cellule costituenti. Tali analisi dei biospecimen di pazienti archiviati sono state precedentemente escluse dall'analisi a causa dei vincoli di input SCRNA-seq. Ora i ricercatori possono applicare "Omics Insights a una gamma più ampia di campioni del mondo reale che influenzano la diagnostica e la terapia di precisione.
In che modo il protocollo snRNA-seq minimizza le perturbazioni trascrizionali?
Un grande vantaggio di snrna-seq risiede nei suoi più delicati protocolli di dissociazione che prevengono i problemi tecnici derivanti dal calore, tempi di incubazione prolungati o trattamenti enzimatici eccessivi. Il brief, Metodo di lisi lieve impiegato per la separazione nucleare impedisce i cambiamenti trascrizionali indotti dallo stress che possono distorcere i risultati di scrNA-seq. Senza sottoporre le cellule intatte a gravi gradi di dissociazione, Il campionamento SnRNA-seq conserva meglio l'integrità delle trascrizioni presenti al momento della dissociazione.
In che modo snRNA-seq minimizza i modelli di espressione genica spuri?
Ciò riduce al minimo il verificarsi di schemi di espressione genica spuriati causati dal processo di dissociazione stesso. I ribosomi maturi trovati esclusivamente all'interno di compartimenti citoplasmatici significano che qualsiasi mRNA che codifica per i geni di risposta allo stress immediato espresso non può essere tradotto. I loro obiettivi trascrizionali a valle rimangono così inalterati. Studi comparativi che applicano entrambe le tecniche allo stesso tessuto hanno dimostrato che snRNA-seq cattura un sottoinsieme più ampio di tipi di cellule con meno perturbazione trascrizionale indotta da dissociazione.
In che modo le durate di lisi brevi aiutano a controllare i pregiudizi trascrittomici in snrna-seq?
Ulteriori fattori come le durate di lisi breve aiutano a controllare i pregiudizi trascrittomici. L'esposizione prolungata sul ghiaccio prima di procedere con la preparazione della libreria a nucleo singolo può avere un impatto negativo sulla qualità dei dati avviando cambiamenti trascrizionali. L'attenta tempistica alletta tali effetti. Sufficienti importi del tessuto iniziale assicurano anche preparati di nuclei di alta qualità necessari per una robusta profilazione dati limitazioni metodologiche.
In che modo i flussi di lavoro SnRNA-Seq ottimizzati minimizzano gli artefatti tecnici?
Complessivamente, I flussi di lavoro SnRNA-Seq ottimizzati minimizzano i fattori pre-analitici che complicano l'interpretazione biologica. La loro delicata estrazione del contenuto nucleare presenta profili di espressione genica modellati meno da artefatti tecnici rispetto ai processi biologici endogeni. Ciò offre una maggiore chiarezza e fiducia alle scoperte raccolte per le reti normative, rare sottopopolazioni, e programmi di specializzazione cellulare all'interno di sistemi complessi in vivo.
In che modo i ricercatori possono applicare snrna-seq per far avanzare la comprensione biologica?
Come tecnica innovativa che perfeziona la nostra visione della complessità cellulare, SnRNA-seq apre nuove frontiere per diverse applicazioni di ricerca. Alcuni esempi evidenziano il suo potenziale per guidare il progresso scientifico:
- Biologia dello sviluppo: Il confronto dei nuclei da fasi embrionali distinte potrebbe rivelare programmi normativi specifici per lo stadio che guidano la differenziazione. Spazialmente risolti snRNA-seq mapping i gradienti di sviluppo possono scoprire i meccanismi di motivi.
- Patologia della malattia: Profilazione malata contro nuclei tissutali sani può identificare i cambiamenti associati alla patologia nei tipi di cellule rari che illuminano i meccanismi. Le analisi del campione dei pazienti potrebbero informare la diagnostica e la terapia di precisione.
- Neuropsichiatria: Indagare sul tessuto cerebrale post mortem archiviato tramite snrna-seq aiuta a caratterizzare la neurodiversità delle condizioni psichiatriche. Gli studi longitudinali possono scoprire biomarcatori longitudinali e bersagli terapeutici.
- Studi evolutivi: SnRNA-seq rende i confronti fattibili delle specie incrociate precedentemente impegnative a causa delle differenze di inter-organismo nelle proprietà dei tessuti. Come “Phylo-snrna-seq” Informa la nostra comprensione dei processi normativi conservati rispetto a quelli divergenti.
- Integrazione multi-omica: L'associazione di snrna-seq con tecniche di profilazione spaziale produce mappe epigenomiche e trascrittomiche risolte spazialmente che chiariscono i microambienti nucleari. L'integrazione dei dati di trascrizione spaziale con strati proteomici o metabolomici fornisce un contesto a livello di sistema.
Riepilogo
Come una delle numerose tecniche emergenti che rivoluzionano l'analisi a cellula singola, snRNA-seq catalizza le scoperte rendendo sperimentalmente trattabili campioni precedentemente intrattabili. Il suo ridimensionamento di omiche nucleari promette nuove intuizioni che portano diversi campi dalla medicina rigenerativa alla precisione oncologia. Insieme ad approcci complementari, Snrna-seq aiuta a svelare affascinante complessità della vita a un livello senza precedenti.