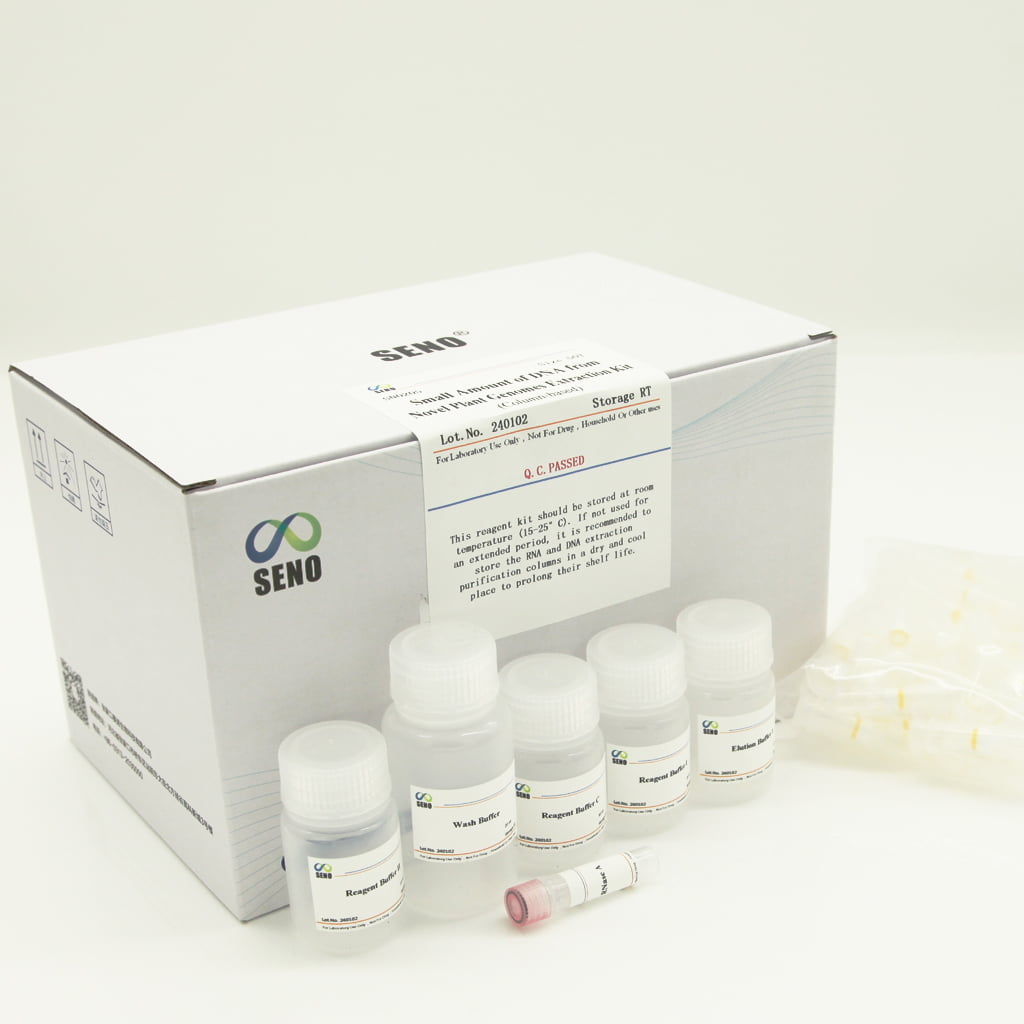
- Componenti del kit di reagenti
| Specifiche | 50T | 100T |
| Gatto. NO. | SN0205 | SN0206 |
| Colonne di estrazione del DNA (impostato) | 50 (impostato) | 100 (impostato) |
| Tampone reagente i | 20 ml | 2×20 ml |
| Tampone reagente II | 15 ml | 15 ml |
| Tampone reagente C | 30 ml | 2 × 30 ml |
| Tampone di eluizione 1 | 15 ml | 2 × 15 ml |
| Tampone di lavaggio | 20 ml | 20 ml |
| RNasi A | 1ml | 1ml |
| Manuale di istruzioni | 1 | 1 |
- Magazzinaggio
Questo kit deve essere conservato a temperatura ambiente (15-25℃) in condizioni asciutte e può essere conservato per 12 mesi. Le colonne di purificazione per l'estrazione del DNA possono essere conservate in un ambiente fresco e asciutto per un massimo di 1 anno. La RNasi A contiene un conservante e può essere trasportata a temperatura ambiente, ma per la conservazione a lungo termine, dovrebbe essere mantenuto a -20 ℃.
- Istruzioni per l'uso del kit di reagenti
3.1 Questo kit è destinato a scopi di ricerca in biologia molecolare e non deve essere utilizzato per la diagnosi o il trattamento di malattie.
3.2 Alcuni componenti del kit contengono sostanze irritanti; è opportuno prendere le dovute precauzioni (come indossare indumenti protettivi e occhiali protettivi).
3.3 L'utilizzo di questo kit richiede apparecchiature aggiuntive come una centrifuga ad alta velocità, bagnomaria (bagno di metallo), miscelatore a vortice, etanolo anidro, azoto liquido, cloroformio, acqua deionizzata sterile, e tubi EP.
- Introduzione al kit di reagenti
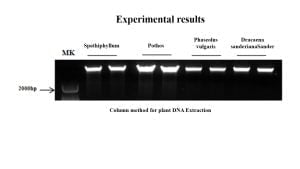
Il nuovo genoma delle piante Purificazione del DNA Il kit fornisce un metodo rapido per la purificazione del DNA. Prepara efficacemente il DNA con tamponi di reagenti specifici II e C, Collezionare DNA di alta purezza attraverso colonne di adsorbimento.
Questo kit è ampiamente applicabile per l'isolamento di campioni da tessuti e funghi vegetali, Abilitare l'estrazione del DNA totale da piante e funghi all'interno 30 minuti. Il processo di estrazione non coinvolge reagenti tossici come il fenolo-cloroformio. Il DNA estratto può essere utilizzato direttamente per esperimenti a valle come PCR e Southern Blotting.
- Principi e procedure sperimentali:

- Processo di estrazione
Precauzioni prima di iniziare l'esperimento:
UN. Tampone reagente I e tampone reagente C tendono a precipitare a basse temperature. Si consiglia di riscaldarli a 65 ° C per 5 minuti fino a quando i precipitati si dissolvono prima dell'uso normale.
B. Prima di usare Tampone di lavaggio 1, Aggiungi la quantità specificata di etanolo assoluto come indicato sull'etichetta della bottiglia di reagente e segna un controllo (√) sull'etichetta per indicare l'aggiunta di etanolo assoluto.
C. Il buffer di eluizione è un 0.1x soluzione TE contenente una quantità minima di EDTA. Se l'EDTA influisce sugli esperimenti successivi, Si consiglia di sostituire il tampone di eluizione con acqua deionizzata sterile.
1. Elaborazione del campione:
UN. Raccolta e stoccaggio dei materiali
Se i materiali appena raccolti non possono essere utilizzati immediatamente, Mettili in azoto liquido per il raffreddamento e finalmente conservare a -80 ° C. I materiali essiccati possono essere conservati a temperatura ambiente.
B. Se le condizioni lo consentono, Raccogli materiali freschi quando possibile, Poiché i materiali freschi contengono meno polisaccaridi e polifenoli.
C. Quando si raccolgono funghi dalla coltura liquida, Separare il liquido e raccogliere le cellule fungine mediante centrifugazione.
2. Pesare intorno 100 mg di campioni freschi o non superanti 20 mg di materiale secco e macinarlo con azoto liquido.
(Nota: Diversi importi del campione possono variare; è consigliabile ottimizzare l'importo del campione attraverso prove pre-sperimentali.)
3. Aggiungere 400tampone reagente μl I e 10 μL di RNASEA (10 mg/ml), Garantire che non ci siano gruppi nel campione di terra. I ciuffi sono difficili da lisare, Ridurre la resa del DNA. Anche, Non mescolare Reagente tampone I e rnasea prima dell'utilizzo.
4. Incubare a 65°C per 5 minuti, invertire delicatamente la miscela 2-3 volte. Questo passaggio viene utilizzato per la lisi cellulare. Se i campioni sono difficili da lisare, estendere il tempo di incubazione, Ma non oltre 30 minuti.
5. Aggiungere 130tampone reagente μl II e mescolare bene, Quindi il ghiaccio-bagliore per 5 minuti (Questo passaggio viene utilizzato per precipitanti polisaccaridi e proteine).
6. Centrifugare il lisato 5 minuti a 14,000 giri/min (20,000× g).
(Nota: Alcuni materiali vegetali possono contenere molte sostanze viscose in questo passaggio, che può tagliare il DNA nei passaggi successivi. Perciò, Lo stato ideale è rimuovere queste sostanze durante questo passaggio. Dopo la centrifugazione, Trasferisci il surnatante in una nuova tuba di centrifuga. Se c'è una quantità significativa di materiale flocculento nel surnatante dopo la centrifugazione, indica che la quantità iniziale del campione era troppo grande. Prendi in considerazione la riduzione dell'importo del campione iniziale.)
7. Trasferire con cura il liquido ottenuto in una nuova tuba di centrifuga.
(Nota: Circa 450 μl di liquido possono essere trasferiti; per alcune specie, Può essere inferiore a 450 μl.)
8. Aggiungere un volume uguale di Tampone reagente C e un volume uguale di etanolo assoluto al lisato e mescolare bene.
(Per esempio: Aggiungi 450μL di tampone reagente C, Quindi aggiungere 450 μl di etanolo assoluto. Se il volume del lisato è inferiore a 450 μl, Ridurre proporzionalmente la quantità di tampone c di reagente. L'aggiunta del tampone reagente C causerà lievi precipitazioni, Ma non influirà sugli esperimenti successivi.)
9. Aggiungi il liquido ottenuto alla colonna di purificazione dell'estrazione del DNA (kit) (circa 650-700μl ogni volta), centrifuga a più di 8,000 giri al minuto per 1 minuto, scartare i rifiuti raccolti, e reinserire il tubo di raccolta nella colonna di purificazione per il passaggio successivo.
10. Ripeti il passaggio 9, Aggiungi il liquido rimanente alla colonna di purificazione dell'estrazione del DNA (kit), centrifuga a più di 8,000 giri al minuto per 1 minuto, scartare i rifiuti e il tubo di raccolta.
11. Posizionare la colonna di purificazione per l'estrazione del DNA (kit) nel tubo di raccolta, aggiungere 300ml di tampone di lavaggio 1,centrifuga a più di 8,000 giri al minuto per 1 minuto, scartare i rifiuti, e posizionare la colonna di purificazione per l'estrazione del DNA (kit) nuovamente nel tubo per il passaggio successivo.
(Nota: Conferma l'aggiunta di etanolo assoluto nel tampone di lavaggio 1.)
12. Aggiungere 500ml di tampone di lavaggio 1 alla colonna di purificazione per l'estrazione del DNA (kit), centrifugare a 14,000 giri/min (20,000× g) per 2 minuti, estendere il tempo di centrifugazione in modo appropriato per garantire che la membrana sia adeguatamente asciutta.
13. Posizionare la colonna di purificazione per l'estrazione del DNA (kit) in una nuova provetta da centrifuga, Lascialo scoperto, e incubare a 65 ° C per 2 minuti. Estendere questo passaggio se necessario per evaporare l'etanolo per impedire ai residui di etanolo di influenzare gli esperimenti a valle.
14. Pipetta 100μl di tampone di eluizione sulla membrana della colonna, centrifugare a 12,000 giri al minuto per 2 minuti.
(Nota: 1. L'uso di 50 μl di tampone di eluizione al DNA eluto può aumentare la concentrazione del DNA ma ridurre la resa complessiva del DNA; 2. L'eluata contenente DNA può essere riapplicato alla colonna di purificazione dell'estrazione del DNA e centrifugato a 12,000 giri al minuto per 2 minuti di nuovo per aumentare la resa del DNA.)







Recensioni
Non ci sono ancora recensioni.