Prosedur QPCR untuk deteksi Chlamydia melibatkan dua langkah utama: Ekstraksi DNA dan amplifikasi dan pemeriksaan DNA berikutnya. Mulanya, Kami akan fokus membagikan konten pembelajaran yang terkait dengan ekstraksi DNA. Artikel ini terutama menggali fondasi teoretis dan pengantar reagen yang digunakan.
Daftar isi
BeralihPrinsip Pemurnian Teoritis Ekstraksi DNA Sebelum Melakukan Eksperimen, Memahami prinsip -prinsip dasar dan prosedur operasional bermanfaat. Ini membantu kita memahami percobaan, meramalkan hasil potensial, dan hindari hanya menjadi operator yang berulang.
Prinsip dasar ekstraksi DNA
Integritas struktur DNA: Misalnya, Prinsip di balik deteksi Chlamydia berbasis penyelidikan QPCR melibatkan deteksi spesifik Chlamydia dengan memperkuat wilayah pengkodean 16srrna dalam genom Chlamydia. Amplifikasi spesifik klamidia dapat dideteksi pada 520nm (Saluran fam). DNA klamidia yang tidak lengkap akan gagal menjalani amplifikasi dan deteksi karakteristik.
Pertimbangan:
(1) Untuk memastikan ini, Prosedur eksperimental kami harus melibatkan penanganan yang lembut untuk mencegah degradasi DNA.
(2) Perhatian harus diberikan untuk menonaktifkan DNAS.
(3) Jika kami hanya membeli kit reagen QPCR Chlamydia tanpa kit reagen ekstraksi yang sesuai, Kami tidak hanya perlu menguji penerapan kit uji tetapi juga memvalidasi efektivitas proses ekstraksi kami.
1.1.2 Kemurnian DNA: Berusaha untuk menghilangkan zat molekul besar lainnya yang mungkin mengganggu DNA, dengan demikian menghindari dampak pada percobaan selanjutnya. Pastikan bahwa sampel yang diekstraksi tidak mengandung pelarut organik atau konsentrasi ion logam yang tinggi yang menghambat enzim.
Pertimbangan:
(1) Pertimbangan serupa berlaku saat membatasi jumlah total sel dalam sampel sel, Karena DNA non-target dalam zat molekul besar lainnya dapat mempengaruhi percobaan.
(2) Catatan peringatan untuk kontrol kualitas (QC): Pengambilan sampel tidak boleh berlebihan. Sambil meningkatkan volume uji mungkin diinginkan untuk keterwakilan di QC, Perhatian harus dilakukan dalam kasus seperti itu.
(3) Penting untuk memahami sampel kami; Jika mengandung pelarut organik atau ion logam yang menghambat enzim, kita perlu mengkonfirmasi apakah zat ini dapat dihilangkan secara efektif sebelum percobaan atau apakah konsentrasinya dapat dikurangi melalui metode kultur sel.
(4) Kontaminasi dari asam nukleat lainnya adalah faktor gangguan yang umum.
Khususnya, Saat menangani chlamydia atau vortex spesimen chlamydia positif, Aerosol dapat dihasilkan. Karena itu, Selama pemeriksaan klamidia, Operasi aseptik yang ketat dalam kabinet biosafety sangat penting, dan sentrifugasi cepat setelah vortexing disarankan.
Langkah Ekstraksi DNA
Ekstraksi DNA hanya melibatkan dua langkah: lisis sel dan pemurnian.
- Lisis sel lisis sel memecah struktur sel sampel, Melepaskan DNA dan komponen seluler lainnya ke dalam larutan. Metode untuk lisis DNA termasuk mekanis, kimia, dan proses enzimatik.
- Metode mekanis sering melibatkan penggilingan nitrogen cair.
- Metode kimia seperti CTAB Dan SDS bertujuan untuk melarutkan selaput sel. CTAB bertindak sebagai pembersih kationik, Sedangkan SDS adalah pembersih anionik, keduanya membantu pembubaran membran sel. Membersihkan protein denature, mengganggu struktur membran dan melepaskan protein yang terhubung ke asam nukleat.
- Aksi enzimatik, seperti pencernaan oleh proteinase k.
Pertimbangan:
(1) Untuk target deteksi produk terapi sel kami, khususnya supernatan sel, Pencernaan enzimatik adalah pendekatan yang lebih lembut dan lebih sederhana.
(2) Aktivitas enzim dipengaruhi oleh faktor lingkungan seperti suhu dan ion garam. Memahami Sampel awal sangat penting untuk menghindari efek potensial dari pelarut organik atau konsentrasi ion garam yang tinggi. Buffer dapat ditambahkan untuk membuat lingkungan latar belakang yang sesuai.
Pemurnian pemurnian DNA memisahkan DNA dari komponen seluler lainnya seperti protein, lipid, polisakarida, dan RNA.
Setelah struktur sel dilisiskan, Pendekatan dasar untuk mengisolasi DNA bergantung pada mengeksploitasi perbedaan sifat fisik dan kimia antara DNA dan komponen lain seperti RNA, protein, dan lipid.
- DNA memiliki kelarutan terendah dalam larutan 0,14mol/L NaCl. Melarutkan DNA dalam larutan konsentrasi garam tinggi dapat menghilangkan kotoran yang tidak larut dalam garam tinggi. DNA mengendap menggunakan larutan garam rendah menghilangkan kotoran yang larut dalam garam rendah.
- Sebagian besar protein tidak toleran terhadap suhu antara 60-80 ℃, sedangkan DNA mendenaturasi di atas 80 ℃.
- DNA tidak larut dalam alkohol, tetapi protein tertentu dalam sel dapat larut di dalamnya.
Pertimbangan:
(1) Memahami poin -poin ini membantu memahami berbagai metode pemurnian.
(2) Saat memverifikasi efektivitas ekstraksi dan pemurnian, modifikasi atau penambahan pada langkah tertentu mungkin diperlukan berdasarkan prinsip eksperimental.
Metode Ekstraksi DNA Umum untuk Pemeriksaan DNA Chlamydia, Memahami metode pemeriksaan arus utama yang ditawarkan oleh produsen kit uji klamidia sangat penting. Metode umum termasuk metode berbasis manik dan berbasis curah hujan. Metode penyaringan, Karena biaya yang lebih tinggi, saat ini tidak dipertimbangkan, sambil mempertimbangkan biaya, Metode kolom centrifuge mungkin merupakan preferensi pribadi.
Metode Presipitasi Metode presipitasi melibatkan ekstraksi fenol/kloroform untuk menghilangkan protein dan etanol/isopropanol untuk mengendapkan DNA. Ekstrak fenol protein terdenaturasi dari fase berair, menghambat degradasi DNA oleh DNAS. Kloroform mempercepat pemisahan fase organik dan berair, Menghapus sisa fenol.
Pertimbangan:
(1) Ketakutan kloroform membuatku merasa tidak nyaman di luar kap asap.
(2) Mempertimbangkan kemampuannya untuk menangani volume sampel yang besar, Saya cenderung mencoba metode ini.
(3) Ada sedikit kekhawatiran tentang apakah metode presipitasi dapat memicu kotoran, Jadi diperlukan penelitian lebih lanjut.
Metode kolom centrifuge
Metode ini melibatkan larutan pengikat/proteinase K yang unik yang dengan cepat memecah struktur sel dan menonaktifkan nuklease seluler. DNA genomik secara selektif menganut membran berbasis silikon di dalam kolom centrifuge dalam keadaan garam tinggi. Melalui serangkaian langkah pencucian dan sentrifugasi yang cepat, Zat penghambat dihilangkan bersama dengan metabolit dan protein sel, meninggalkan DNA genom yang dimurnikan, yang kemudian dielusi dari membran berbasis silikon menggunakan buffer elusi rendah garam.
Pertimbangan:
(1) Cukup aman, Tapi volume sampel mungkin agak rendah.
(2) Volume terbatas tabung pengumpulan membatasi ruang operasional.
(3) Untuk memastikan deteksi, DNA perlu terkonsentrasi, yang selanjutnya mengurangi volume pipetting. Menguasai pipet sangat penting.
(4) Mempertimbangkan biaya, Metode kolom centrifuge adalah pilihan sementara.
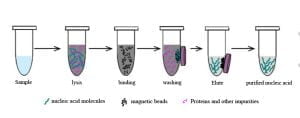
Metode manik magnetik
Langkah -langkah utama dari metode manik magnetik melibatkan lisis, mengikat, pencucian, dan elusi. Langkah -langkah spesifik tidak dirinci di sini.

Buffer lisis yang digunakan dalam metode ini mendenaturasi protein, menyebabkan lisis sel dan denaturasi protein yang terikat pada DNA. Manik -manik magnetik secara khusus menyerap DNA. Melalui mencuci, kotoran seperti RNA dan protein, Terlepas dari DNA, dihapus. Kemudian, Solusi elusi memisahkan DNA yang terikat pada manik -manik, menghasilkan DNA yang sangat murni dan terkonsentrasi untuk PCR amplifikasi.
Metode manik magnetik menggabungkan nanoteknologi dan bioteknologi, Menggunakan nanopartikel magnetik terutama terdiri dari bahan aktif biologis yang menunjukkan paramagnetisme. Manik -manik ini memiliki lapisan permukaan yang mampu menjalani pertukaran kation, memfasilitasi adsorpsi asam nukleat. Bahan permukaan mengikat dengan asam nukleat melalui ikatan hidrogen dan interaksi elektrostatik. Prinsip pemurnian melibatkan pengikat asam nukleat dalam kondisi garam tinggi dan melukainya di lingkungan rendah garam. Lebih-lebih lagi, manik -manik magnetik agregat atau disebarkan dalam kondisi medan magnet, menghilangkan kebutuhan untuk operasi manual seperti sentrifugasi.
Pertimbangan:
(1) Inti dari metode manik magnetik terletak pada manik -manik. Beberapa kit berisi sejumlah manik -manik, Minta para ahli untuk membangun sistem mereka sendiri. Menantikan pencapaian mereka.
(2) Saat memilih dudukan magnetik, Pilih yang terlihat.
Penilaian Kemurnian DNA
Setelah ekstraksi DNA, mengevaluasi kemurnian, konsentrasi, dan integritas sangat penting. Metode umum meliputi:
- Elektroforesis gel: Metode ini menggunakan agarosa gel untuk menentukan residu RNA, protein, fenol, atau metabolit lain dalam DNA yang diekstraksi. Selain itu, Kecerahan band menunjukkan integritas DNA. Elektroforesis gel, dengan membandingkan kecerahan pita dengan penanda DNA, secara kasar memperkirakan konsentrasi DNA.
- Spektrofotometri: Cincin benzena basa DNA atau RNA sangat menyerap dalam kisaran ultraviolet pada 260nm. Protein menunjukkan penyerapan maksimum pada 280nm, Sementara garam dan molekul kecil berkonsentrasi pada 230nm. Menggunakan spektrofotometer, Prinsipnya adalah bahwa absorbansi berkorelasi dengan konsentrasi DNA. Menilai kemurnian DNA biasanya melibatkan pengukuran OD260, OD280, dan nilai OD230, menilai kemurnian berdasarkan rasio mereka.
Biasanya, DNA murni memiliki rasio OD260/OD280 1.8. Rasio di atas 1.9 menyarankan degradasi DNA atau kontaminasi RNA, sementara di bawah 1.7 menunjukkan sisa -sisa protein. Elektroforesis gel sering dilakukan pada awalnya untuk menilai kemurnian dan integritas, diikuti oleh spektrofotometri UV untuk mengukur konsentrasi.
Metode Deteksi Qbit
Teknik ini menggunakan pewarna fluorescent yang memancarkan fluoresensi setelah mengikat molekul target tertentu. Instrumen mengukur nilai fluoresensi dan mengubahnya menjadi data konsentrasi. Dibandingkan dengan spektrofotometri, Metode ini lebih sensitif, spesifik, dan tepat dalam deteksi.
Pertimbangan:
(1) Mempertimbangkan biaya, metode 1.5.1 Dan 1.5.2 saat ini lebih nyaman selama pengembangan metode ekstraksi.
(2) Untuk mengkonfirmasi efektivitas ekstraksi, Langkah -langkah kontrol kualitas selanjutnya diperlukan. Saat mengembangkan metode secara mandiri, Memilih kontrol kualitas yang efektif sangat penting.
Pengantar Informasi Ekstraksi DNA dan Pereaksi Pemurnian
Saya telah menyusun ringkasan dari berbagai reagen yang mungkin digunakan untuk pemahaman yang lebih baik. Kompilasi ini untuk tujuan referensi, tidak asli, semata -mata dimaksudkan untuk pertukaran pendidikan.
Proteinase k proteinase k, terisolasi dari jamur putih, adalah enzim pencernaan protein yang kuat dengan aktivitas spesifik yang tinggi, penting untuk ekstraksi DNA. Itu tetap aktif dalam kisaran pH yang luas (4-12.5) dan pada suhu tinggi (50-70 Derajat Celcius). Agen pengkelat seperti EDTA atau agen deionisasi seperti SDS tidak menonaktifkannya.
Proteinase K, protease serin dengan aktivitas pembelahan yang luas, memotong ikatan peptida di ujung karboksil asam amino alifatik dan aromatik. Ini mencerna berbagai protein membran dan glikoprotein pada membran sel dan nuklir dan juga mencerna histone yang terikat pada DNA.
Natrium klorida (NaCl) NaCl membantu dalam menghilangkan protein yang terikat pada DNA. Dengan menetralkan muatan negatif pada DNA, Ini memungkinkan molekul untuk menggabungkan dan membuat protein larut dalam lapisan air, mencegah presipitasi mereka dengan DNA dalam alkohol.
Konsentrasi garam memainkan peran penting ketika sel terpapar solusi hipotonik atau hipertonik, mengarah ke osmosis.
Edta edta chelates kation divalent seperti mg2+ dan ca2+, yang ada dalam enzim, Mengurangi aktivitas enzimatik DNAS dan RNAS. Misalnya, Enzim DNase membutuhkan ion Mg2+ sebagai kofaktor untuk aktivitasnya. Ion Chelating Mg2+ dengan EDTA membuat DNase tidak aktif, melindungi DNA. Ion magnesium juga diperlukan untuk agregasi asam-protein nukleat, Sementara ion kalsium sangat penting untuk integritas dinding sel dan stabilitas membran.
Fenol, pelarut organik tidak larut dalam air, digunakan dengan kloroform dan alkohol isoamil untuk pemurnian DNA, Menghapus protein dan kontaminan polisakarida. Saat diguncang dengan ekstrak seluler, Komponen sel nonpolar akan fraksinasi dalam fenol, Meninggalkan komponen kutub di dalam air. DNA tetap tidak terpecahkan dalam fenol karena itu adalah pelarut nonpolar. Di sisi lain, Protein mengandung kedua kelompok polar dan nonpolar karena asam amino yang berbeda dalam rantai panjangnya. Lipatan protein ke sekunder, tersier, dan struktur kuaterner juga tergantung pada polaritas asam amino.
Sentrifugasi dengan campuran fenol: dan kloroform:
alkohol isoamil dalam a 25:24:1 Rasio menghasilkan tiga lapisan: encer, interphase, dan organik. DNA, bermuatan negatif dan polar pada pH netral ke alkali, tetap hidrofilik dan dipertahankan dalam fase air. Asam amino hidrofobik dalam protein melindungi DNA dalam larutan berair. Namun, setelah denaturasi, asam nukleat nonpolar terpapar, menyebabkan protein dan polisakarida tertentu mengendap di interphase.
Kombinasi fenol-kloroform mengurangi distribusi poli(A) dan mRNA dalam fase organik dan meminimalkan pembentukan kompleks RNA-protein yang tidak larut di interfase. Fenol mempertahankan sekitar 10-15% dari fase berair, mengakibatkan kerugian RNA yang serupa. Kloroform mencegah retensi air, dengan demikian meningkatkan hasil.
Hanya fenol netral yang harus digunakan sebagai fenol asam melarutkan DNA, atau fenol, melalui aksi oksidatif, berubah menjadi kuinon, menghasilkan radikal bebas yang menurunkan asam nukleat.
Khloroform
Kloroform adalah nonpolar (hidrofobik) pelarut di mana protein dan lipid nonpolar larut. Ini mendorong distribusi lipid dan puing -puing seluler ke dalam fase organik, melindungi DNA yang terpisah dalam fase air. Dengan kepadatan yang relatif tinggi (1.47 g/cm³), Kloroform memastikan pemisahan kedua cairan, memungkinkan fase organik dan berair untuk secara jelas terpisah dan membantu menghilangkan fase air sambil meminimalkan kontaminasi silang dengan fase organik. Karena kloroform secara inheren volatile, itu tidak menghalangi proses selanjutnya.
Suplemen: Cara menggunakan fenol dan kloroform untuk ekstraksi DNA untuk menghilangkan protein?
Fenol dan kloroform adalah molekul nonpolar, Sedangkan air adalah molekul kutub. Saat larutan protein-air bercampur dengan fenol atau kloroform, Molekul air antara molekul protein dipindahkan oleh fenol atau kloroform, menyebabkan protein kehilangan keadaan terhidrasi dan denature. Setelah sentrifugasi, protein terdenaturasi, lebih padat dari air, terpisah dari fase berair dan mengendap di bawahnya, memisahkan dari DNA yang dilarutkan dalam fase berair. Fenol dan kloroform, menjadi pelarut organik, memiliki kelebihan dan kekurangan dalam penghapusan protein. Fenol memiliki efek denaturasi yang lebih signifikan tetapi sebagian larut dalam fase berair, mengakibatkan hilangnya 10% ke 15% DNA dalam fase air. Efek denaturasi kloroform tidak seefektif fenol, Tapi itu tidak larut dalam air, karenanya tidak membawa DNA. Dengan demikian, Kombinasi fenol dan kloroform menghasilkan hasil terbaik selama proses ekstraksi.
Isopentanol (Isopropanol)
Kloroform yang terpapar udara membentuk gas berbahaya, Phosgene. Kalau saja kloroform digunakan, perangkap gas menyebabkan berbusa atau menggelegak, membuatnya sulit untuk memurnikan DNA dengan benar antara kedua fase. Pada titik ini, Menggabungkan kloroform dengan isopropanol atau octanol mencegah emulsi larutan selama proses ekstraksi.
RNase A
RNase A adalah endoribonuklease yang mengkatalisasi hidrolisis 3′,5′-Ikatan Fosfodiester RNA di 5′-ikatan ester dalam reaksi dua langkah. Langkah pertama adalah transfosforilasi, mengakibatkan penghentian oligonukleotida yang berakhir dengan pirimidin 2′,3′-fosfat siklik. Langkah kedua adalah hidrolisis siklik fosfat untuk menghasilkan terminal 3-fosfodiester. DNA tidak memiliki yang penting 2′-OH, membuatnya tahan terhadap pencernaan oleh RNase a.
Isopropanol/etanol
Etanol memicu DNA dari larutan ekstraksi. DNA, dengan tulang punggung fosfodiester, pada dasarnya hidrofilik. Molekul air membentuk cangkang hidrasi di sekitar DNA melalui ikatan hidrogen. Isopropanol/etanol digunakan untuk mengendapkan DNA, mengganggu cangkang hidrasi. Isopropanol adalah pilihan yang baik untuk presipitasi DNA. Itu membutuhkan volume yang lebih kecil (0.6–0.7 kali volume supernatan). RNA, dengan tambahan 2'oh, cenderung lebih larut dalam air dan diendapkan secara selektif, meninggalkan DNA. Isopropanol dapat melarutkan pelarut nonpolar seperti kloroform, dengan demikian menghilangkan kotoran dari langkah sebelumnya.
Khas, isopropanol dingin digunakan, Tetapi para peneliti menyarankan suhu kamar untuk mencegah curah hujan polisakarida. Sementara suhu rendah meningkatkan hasil DNA, Mereka mungkin meningkatkan kotoran.
Isopropanol (Keuntungan: membutuhkan lebih sedikit volume, curah hujan cepat, Cocok untuk konsentrasi rendah, Sampel DNA volume tinggi; Kerugian: rentan terhadap garam copresipitating dengan DNA; membutuhkan banyak pencucian dengan 70% etanol) Etanol (Keuntungan: presipitasi minimal dengan garam, Etanol residual dalam presipitasi DNA dengan mudah menguap, tidak mempengaruhi percobaan selanjutnya; Kerugian: volume keseluruhan yang lebih besar, Membutuhkan penyimpanan berkepanjangan pada -20 ° C untuk curah hujan yang efektif, memerlukan 70% Cuci etanol)
Sodium asetat/amonium asetat/kalium asetat/natrium klorida/lithium klorida/kalium klorida
Garam dalam prosedur ekstraksi berfungsi dengan menetralkan muatan pada tulang punggung gula-fosfat DNA. Sodium asetat pada pH 5.2 biasanya digunakan bersama etanol untuk presipitasi asam nukleat. Dalam solusi, natrium asetat terdisosiasi menjadi Na+ dan [Ch3coo]- ion. Ion natrium bermuatan positif menetralkan muatan negatif pada PO3- dari tulang punggung gula-fosfat asam nukleat, mengurangi tolakan antara molekul DNA. Ini secara signifikan mengurangi hidrofilisitas molekul DNA, karenanya sangat mengurangi kelarutan mereka di dalam air.
Etanol
Cuci DNA endapan dengan 70% etanol untuk menghilangkan garam berlebih, alat sentrifugal, dan membuang etanol, meninggalkan DNA dalam endapan. Keringkan udara atau vakum endapan. Hindari pengeringan berlebihan karena ini dapat membuatnya sulit untuk melarutkan DNA nanti.
Tris-edta (Itu) Buffer/air steril
Dalam metode isolasi DNA sebelumnya, DNA perlu dikeringkan dan disimpan, Kemudian diencerkan sesuai kebutuhan. Dewasa ini, untuk penyimpanan jangka panjang, lebih bijaksana untuk menyimpan DNA dalam buffer yang mempertahankan pH dan mencegah degradasi. TE Buffer berisi Tris (10 mm) dan edta (1 mm), Di mana Tris bertindak sebagai buffer dan EDTA sebagai agen pengkelat. Untuk isolasi DNA, pH biasanya diatur ke 7.5-8.5. Alkalinitas yang lemah dari buffer TE membantu mencegah kemungkinan hidrolisis asam, lebih lanjut menstabilkan DNA dalam air.
Komponen Tris Amino dalam TE Buffer melindungi rantai DNA dari kerusakan radiasi dalam larutan padat dan cair. Saat radiasi menghasilkan radikal bebas, itu dapat membahayakan rantai DNA. Dengan demikian, dalam solusi cairan pada suhu kamar, Tris bertindak dengan membersihkan radikal bebas hidroksil.
EDTA bertujuan untuk mengubah ion Mg2+ dalam solusi yang diperlukan untuk aktivitas DNase atau RNase, Melindungi DNA dari serangan DNase atau RNase.
Air steril dapat digunakan untuk penyimpanan DNA jangka pendek. Jika Buffer TE digunakan untuk penyimpanan DNA, Air steril harus lebih jauh melemahkannya untuk mengurangi konsentrasi EDTA. Ini memastikan ion magnesium tersedia untuk aktivitas polimerase selama PCR. Komponen penyangga dalam te mungkin menghambat amplifikasi DNA jika DNA perlu diurutkan nanti.