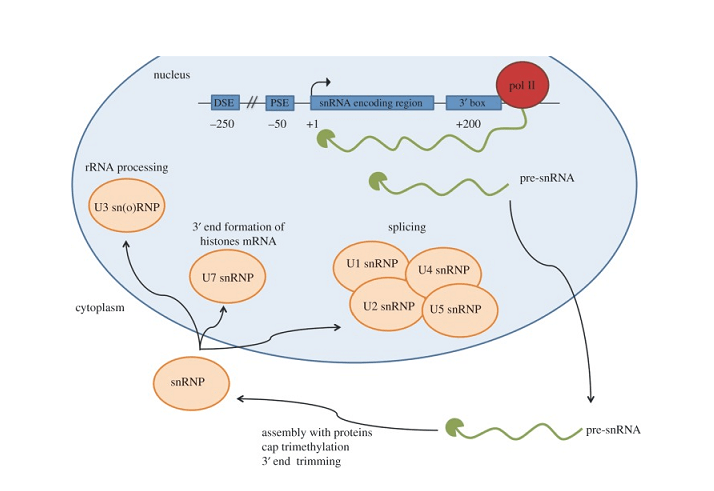
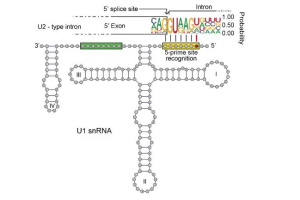
ARN nucleares individuales, o snRNA, Desempeñan un papel crucial dentro de las células a pesar de su corta longitud.. Como moléculas de ARN no codificantes que se encuentran únicamente en el núcleo., Los snRNA participan en modificaciones postranscripcionales esenciales que ayudan a sintetizar proteínas funcionales.. Hay dos clases principales de snRNA. – U1, U2, U4, U5, y U6 que forman las pequeñas ribonucleoproteínas nucleares. (snRNP) involucrado en el empalme, y los snoRNA de caja H/ACA y C/D que guían las modificaciones químicas de otros ARN.
¿Qué es la secuenciación de ARN de un solo núcleo? (snRNA-seq)?
Secuenciación de ARN de un solo núcleo, o snRNA-seq, es una técnica innovadora que analiza el transcriptoma nuclear aislado de células individuales. Este enfoque ayuda a minimizar las respuestas de estrés transcripcional que pueden distorsionar los resultados durante la disociación.. En comparación con la secuenciación tradicional de ARN unicelular (secuencia de ARNc), snRNA-seq exhibe ciertos beneficios que mejoran su utilidad para investigar la heterogeneidad celular.
¿Cuál es la diferencia entre snRNA-seq y scRNA-seq??
Secuenciación de ARN de un solo núcleo (snRNA-seq), también conocido como snRNA-seq o sNuc-seq, es un método innovador de secuenciación de ARN que se utiliza para perfilar la expresión genética en células que son difíciles de aislar. Esta técnica constituye una poderosa alternativa a la secuenciación de ARN unicelular (secuencia de ARNc) analizando núcleos en lugar de células intactas. Pero, ¿qué diferencia exactamente a snRNA-seq de scRNA-seq?? Profundicemos en las diferencias clave.
scRNA-seq mide transcripciones tanto citoplásmicas como nucleares, Proporcionar una visión completa del perfil de expresión genética de una célula.. En contraste, snRNA-seq mide principalmente transcripciones nucleares, ya que se centra en analizar el ARN dentro del propio núcleo. Este enfoque dirigido minimiza la aparición de expresión genética espuria., Los ribosomas maduros residen en el citoplasma.. Por lo tanto, cualquier factor de transcripción expresado después del proceso de disociación no se puede traducir, impidiendo la transcripción de sus objetivos posteriores.
¿Cómo se adapta snRNA-seq a tipos de tejidos desafiantes??
por uno, snRNA-seq se adapta a una variedad más amplia de tipos de tejidos cuya disociación completa en células individuales puede resultar difícil. Tejidos complejos con matrices extracelulares densas como el cerebro., riñón, y el corazón se puede analizar de forma más eficaz utilizando snRNA-seq debido a su protocolo de disociación más suave. Esto preserva la integridad de los tipos de células sensibles que pueden sufrir cambios transcripcionales o degradarse durante una disociación más dura requerida para scRNA-seq..
¿Cuáles son los beneficios de utilizar snRNA-seq para muestras archivadas??
SnRNA-seq también apoya el estudio de muestras clínicas archivadas. Los tejidos congelados o fijados que serían incompatibles con scRNA-seq se pueden perfilar siempre que se puedan aislar núcleos intactos. Esto amplía el alcance de las posibles aplicaciones de investigación en comparación con otros métodos de secuenciación que exigen células individuales recién aisladas.. Notablemente, snRNA-seq ha permitido a los investigadores analizar tejido cerebral humano post mortem para descubrir la diversidad neuronal a nivel unicelular.
¿Qué perspectiva única proporciona snRNA-seq??
En lugar de una visión integral del transcriptoma completo, snRNA-seq proporciona información centrada en los procesos nucleares y las interacciones moduladas por snRNA. Ofrece una perspectiva excepcionalmente ampliada sobre los mecanismos regulatorios dentro del espacio nuclear confinado.. La aplicación de snRNA-seq a muestras de tejido complejas ha revelado nuevos tipos y estados de células que pueden pasar desapercibidos utilizando scRNA-seq solo. Estos descubrimientos profundizan nuestra comprensión de la organización y especialización celular., ampliando las fronteras de la medicina de precisión.
¿Cómo puede snRNA-seq ayudar a perfilar la expresión genética en muestras desafiantes??
Las capacidades de creación de perfiles nucleares de snRNA-seq resultan especialmente valiosas para muestras que son problemáticas de analizar utilizando enfoques convencionales de scRNA-seq.. Ciertos tipos de tejidos plantean dificultades técnicas debido a cualidades como la composición densa de la matriz., fragilidad ante la disociación, o incompatibilidad con los tratamientos enzimáticos necesarios para el aislamiento unicelular. Las muestras archivadas conservadas mediante fijación también presentan desafíos..
¿Cómo se utilizó snRNA-seq para estudiar el tejido cardíaco de mamíferos??
El tejido cardíaco de mamífero ejemplifica un sistema intratable a scRNA-seq pero susceptible al análisis de snRNA-seq. Las células del músculo cardíaco forman una intrincada red interconectada que no se interrumpe fácilmente sin daño celular. Sin embargo, en 2020 Investigadores alemanes secuenciaron con éxito el primer corazón de mamífero adulto utilizando snRNA-seq. Aislando núcleos en lugar de cardiomiocitos completos, Obtuvieron distribuciones prácticas de poblaciones celulares distintivas dentro del miocardio.. Tal elaboración de perfiles a escala no sería práctica mediante la metodología scRNA-seq.
¿Cómo beneficia snRNA-seq al estudio de poblaciones neuronales??
Las poblaciones neuronales representan otra clase que se beneficia de las capacidades de snRNA-seq. El cerebro está formado por neuronas intrincadamente conectadas que no se disocian fácilmente como células individuales intactas.. Intentar hacerlo puede inducir respuestas de estrés que sesguen los resultados. Sin embargo, el estudio de la heterogeneidad transcriptómica de los subtipos neuronales contribuye a nuestra comprensión de la neurobiología., mecanismos de enfermedad, y más. Los protocolos SnRNA-seq optimizados para tejido del SNC preservan muestras de cerebro humano tanto archivadas como recién resecadas en una resolución unicelular.
¿Cómo permite snRNA-seq el análisis de muestras clínicas congeladas??
Las muestras clínicas congeladas también plantean limitaciones superadas por snRNA-seq. Los estudios de enfermedades hepáticas que compararon núcleos pulmonares sanos y fibróticos detectados mediante snRNA-seq revelaron una identificación imparcial de los tipos de células constituyentes. Dichos análisis de muestras biológicas de pacientes archivadas se excluyeron previamente del análisis debido a limitaciones de entrada de scRNA-seq.. Ahora los investigadores pueden aplicar conocimientos ómicos a una gama más amplia de muestras del mundo real que influyen en el diagnóstico y la terapia de precisión..
¿Cómo minimiza el protocolo snRNA-seq las perturbaciones transcripcionales??
Una ventaja importante de snRNA-seq radica en sus protocolos de disociación más suaves que previenen los problemas técnicos derivados del calor., tiempos de incubación prolongados o tratamientos enzimáticos excesivos. el breve, El método de lisis suave empleado para la separación nuclear previene los cambios transcripcionales inducidos por el estrés que pueden sesgar los resultados de scRNA-seq.. Sin someter a las células intactas a duros pasos de disociación, El muestreo de snRNA-seq preserva mejor la integridad de las transcripciones presentes en el momento de la disociación.
¿Cómo minimiza snRNA-seq los patrones de expresión de genes espurios??
Esto minimiza la aparición de patrones de expresión genética falsos causados por el propio proceso de disociación.. Los ribosomas maduros que se encuentran únicamente dentro de los compartimentos citoplasmáticos significan que cualquier ARNm que codifique genes de respuesta al estrés temprano inmediato expresado después de la disociación no se puede traducir.. Por tanto, sus objetivos transcripcionales posteriores permanecen inalterados.. Los estudios comparativos que aplican ambas técnicas al mismo tejido han demostrado que snRNA-seq captura un subconjunto más amplio de tipos de células con menos perturbación transcripcional inducida por la disociación..
¿Cómo ayudan las duraciones cortas de lisis a controlar los sesgos transcriptómicos en snRNA-seq??
Factores adicionales como las duraciones cortas de la lisis ayudan a controlar los sesgos transcriptómicos. La exposición prolongada al hielo antes de continuar con la preparación de la biblioteca de un solo núcleo puede afectar negativamente la calidad de los datos al iniciar cambios transcripcionales. Una sincronización cuidadosa mitiga tales efectos. Cantidades iniciales suficientes de tejido también garantizan preparaciones de núcleos de alta calidad necesarias para un perfil sólido dadas las limitaciones metodológicas..
¿Cómo los flujos de trabajo optimizados de snRNA-seq minimizan los artefactos técnicos??
En general, Los flujos de trabajo optimizados de snRNA-seq minimizan los factores preanalíticos que complican la interpretación biológica.. Su suave extracción de contenido nuclear presenta perfiles de expresión genética moldeados menos por artefactos técnicos que por procesos biológicos endógenos.. Esto aporta mayor claridad y confianza a los descubrimientos obtenidos sobre las redes regulatorias., subpoblaciones raras, y programas de especialización celular dentro de sistemas complejos in vivo.
¿Cómo pueden los investigadores aplicar snRNA-seq para avanzar en la comprensión biológica??
Como técnica innovadora que refina nuestra visión de la complejidad celular, snRNA-seq abre nuevas fronteras para diversas aplicaciones de investigación. Algunos ejemplos destacan su potencial para impulsar el progreso científico.:
- Biología del desarrollo: La comparación de núcleos de distintas etapas embrionarias podría revelar programas regulatorios específicos de la etapa que guían la diferenciación. Los gradientes de desarrollo del mapeo de snRNA-seq resueltos espacialmente pueden descubrir mecanismos de modelado.
- patología de la enfermedad: Perfilar los núcleos de tejido enfermo versus sano puede identificar cambios asociados a patología en tipos de células raras que iluminan los mecanismos de la enfermedad. Los análisis de muestras de pacientes podrían informar diagnósticos y terapias de precisión.
- Neurosiquiatría: La investigación del tejido cerebral post mortem archivado mediante snRNA-seq ayuda a caracterizar la neurodiversidad de las afecciones psiquiátricas. Los estudios longitudinales pueden descubrir biomarcadores longitudinales y objetivos terapéuticos.
- Estudios evolutivos: SnRNA-seq hace factibles las comparaciones entre especies que antes eran un desafío debido a las diferencias entre organismos en las propiedades de los tejidos. Semejante “filo-snRNA-seq” informa nuestra comprensión de los procesos regulatorios conservados versus divergentes.
- Integración multiómica: El emparejamiento de snRNA-seq con técnicas de perfiles espaciales produce mapas epigenómicos y transcriptómicos resueltos espacialmente que aclaran los microambientes nucleares. La integración de datos de transcripción espacial con capas proteómicas o metabolómicas proporciona un contexto a nivel de sistemas.
Resumen
Como una de varias técnicas emergentes que revolucionan el análisis unicelular, snRNA-seq cataliza descubrimientos al hacer que muestras previamente intratables sean experimentalmente tratables. Su ampliación de la ómica nuclear promete nuevos conocimientos que permitirán avanzar en diversos campos, desde la medicina regenerativa hasta la oncología de precisión.. Junto con enfoques complementarios, snRNA-seq ayuda a desentrañar la fascinante complejidad de la vida a un nivel sin precedentes.