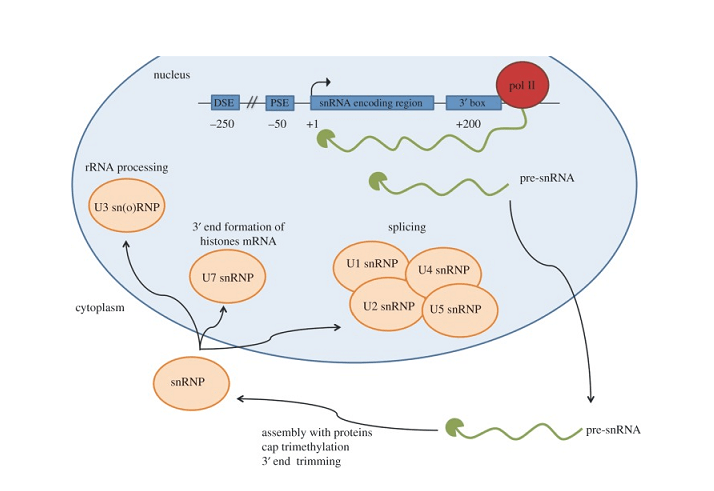
Single Nuclear RNAs, oder snrnas, spielen trotz ihrer kurzen Längen eine entscheidende Rolle in den Zellen. Als nicht kodierende RNA-Moleküle, die ausschließlich im Kern gefunden wurden, SNRNAs nehmen an wesentlichen posttranskriptionellen Modifikationen teil, mit denen funktionelle Proteine synthetisiert werden können. Es gibt zwei Hauptklassen von SNRNAs – U1, U2, U4, U5, und U6, die die kleinen nuklearen Ribonukleoproteine bilden (snrnps) am Spleißen beteiligt, und die H/ACA- und C/D -Box -Snornas, die chemische Modifikationen anderer RNAs leiten.
Was ist eine RNA-Sequenzierung von Einzelnukleus (snrna-seq)?
Einzelnukleus-RNA-Sequenzierung, oder snrna-seq, ist eine innovative Technik, die das aus einzelnen Zellen isolierte Kerntranskriptom analysiert. Dieser Ansatz minimiert Transkriptionsstressreaktionen, die die Ergebnisse während der Dissoziation verzerren können. Im Vergleich zur herkömmlichen Einzelzell-RNA-Sequenzierung (Scnna-seq), SNRNA-seq weist bestimmte Vorteile auf, die ihren Nutzen zur Untersuchung der zellulären Heterogenität verbessern.
Was ist der Unterschied zwischen snrna-seq und scrna-seq?
Einzelnukleus-RNA-Sequenzierung (snrna-seq), auch als snrna-seq oder snuc-seq bezeichnet, ist eine innovative RNA -Sequenzierungsmethode, mit der die Genexpression in Zellen profiliert wird, die zu isolieren sind. Diese Technik dient als leistungsstarke Alternative zur Einzelzell-RNA-Sequenzierung (Scnna-seq) durch Analyse der Kerne anstelle von intakten Zellen. Aber was genau snrna-seq von scrna-seq unterscheidet? Lassen Sie uns die wichtigsten Unterschiede eintauchen.
SCRNA-seq misst sowohl zytoplasmatische als auch nukleare Transkripte, Bereitstellung einer umfassenden Sicht auf das Genexpressionsprofil einer Zelle. Im Gegensatz, SNRNA-seq misst in erster Linie Kerntranskripte, Da es sich auf die Analyse der RNA im Kern selbst konzentriert. Dieser gezielte Ansatz minimiert das Auftreten einer falschen Genexpression, als reife Ribosomen wohnen im Zytoplasma. daher, Alle nach dem Dissoziationsprozess ausgedrückten Transkriptionsfaktoren können nicht übersetzt werden, Verhinderung der Transkription ihrer nachgeschalteten Ziele.
Wie bietet SnRNA-seq anspruchsvolle Gewebetypen??
Für einen, SNRNA-seq berücksichtigt eine größere Vielfalt von Gewebetypen, die möglicherweise schwierig sein können, sich vollständig in einzelne Zellen zu dissoziieren. Komplexe Gewebe mit dichten extrazellulären Matrizen wie dem Gehirn, Niere, und Herz kann aufgrund seines milderen Dissoziationsprotokolls effektiver unter Verwendung snRNA-seq analysiert werden. Dies bewahrt die Integrität sensibler Zelltypen, die Transkriptionsänderungen unterzogen werden oder während der für scra-seq erforderlichen härteren Dissoziation abgebaut werden können.
Was sind die Vorteile der Verwendung von snRNA-seq für archivierte Proben?
SNRNA-seq unterstützt auch die Untersuchung archivierter klinischer Proben. Gefrorene oder feste Gewebe, die mit SCRNA-seq nicht kompatibel wären, kann profiliert werden, solange intakte Kerne isoliert werden können. Dies erweitert den Umfang möglicher Forschungsanwendungen im Vergleich zu anderen Sequenzierungsmethoden, die frisch isolierte Einzelzellen erfordern. Vor allem, SNRNA-seq hat es den Forschern ermöglicht, das menschliche Hirngewebe nach dem Mortem zu analysieren, um die neuronale Vielfalt auf einzelnen Zellen aufzudecken.
Welche einzigartige Perspektive bietet snrna-seq?
Und nicht eine umfassende Sicht auf das vollständige Transkriptom, SNRNA-seq liefert fokussierte Einblicke in nukleare Prozesse und Wechselwirkungen, die von SNRNAs moduliert werden. Es bietet eine einzigartig vergrößerte Perspektive auf regulatorische Mechanismen innerhalb des engen Kernraums. Das Anwenden von snRNA-seq auf komplexe Gewebeproben hat neue Zelltypen und Zustände ergeben, die möglicherweise mit SCRNA-Seq allein unentdeckt bleiben. Solche Entdeckungen vertiefen unser Verständnis der zellulären Organisation und Spezialisierung, Erweiterung der Grenzen der Präzisionsmedizin.
Wie kann snrna-seq dazu beitragen, die Genexpression in herausfordernden Proben zu profilieren?
Die Kernprofilierungsfähigkeiten von snRNA-seq erweisen sich besonders für Proben, die problematisch mit konventionellen SCRNA-Seq-Ansätzen analysieren können. Bestimmte Gewebetypen stellen aufgrund von Qualitäten wie einer dichten Matrixzusammensetzung technische Schwierigkeiten auf, Zerbrechlichkeit bei Dissoziation, oder Inkompatibilität mit enzymatischen Behandlungen, die für die Einzelzellen-Isolierung erforderlich sind. Archivierte Proben, die durch Fixierung erhalten sind, stellen auch Herausforderungen vor.
Wie wurde SNRNA-seq verwendet, um Säugetierherzgewebe zu untersuchen??
Säugetier Herzgewebe veranschaulicht ein System, das für scrna-seq, die dennoch für die snRNA-seq-Analyse zugänglich ist. Herzmuskelzellen bilden ein kompliziertes miteinander verbundenes Netzwerk, das ohne zelluläre Schäden nicht leicht gestört wird. Jedoch, In 2020 Deutsche Forscher haben das erste erwachsene Säugetierherz mit snrna-seq erfolgreich sequenziert. Durch Isolieren von Kernen anstelle von ganzen Kardiomyozyten, Sie erhielten praktische Verteilungen von charakteristischen Zellpopulationen im Myokard. Eine solche Profilerstellung im Maßstab wäre durch die SCRNA-Seq-Methodik unpraktisch.
Wie profitiert snrna-seq der Studie von neuronalen Populationen??
Neuronale Populationen repräsentieren eine andere Klasse, die von snrna-seq-Funktionen profitiert. Das Gehirn besteht aus aufwandend verkabelten Neuronen, die nicht leicht als intakte einzelne Zellen dissoziiert werden. Der Versuch, dies zu tun. Untersuchung der transkriptomischen Heterogenität neuronaler Subtypen beeinflusst jedoch unser Verständnis der Neurobiologie, Krankheitsmechanismen, und mehr. SNRNA-Seq-Protokolle, die für das ZNS-Gewebe optimiert wurden.
Wie aktiviert SnRNA-seq die Analyse von gefrorenen klinischen Proben??
Gefrorene klinische Proben posieren ähnlich Einschränkungen, die durch snrna-seq überwunden werden. Studien zur Lebererkrankung, in denen gesunde und fibrotische Lungenkernen verglichen wurden, die über snRNA-seq nachgewiesen wurden. Solche Analysen archivierter Patienten-Biospecimenen wurden zuvor aufgrund von SCRNA-Seq-Eingangsbeschränkungen ausgeschlossen. Jetzt können sich Forscher bewerben ‚Omics-Erkenntnisse in eine breitere Palette von realen Proben, die die Präzisionsdiagnostik und Therapie beeinflussen.
Wie minimiert das SNRNA-Seq-Protokoll Transkriptionsstörungen??
Ein wesentlicher Vorteil von snrna-seq liegt in seinen sanfteren Dissoziationsprotokollen, die technische Probleme aus der Hitze verhindern, verlängerte Inkubationszeiten oder übermäßige enzymatische Behandlungen. Der Brief, Eine milde Lyse-Methode zur Kerntrennung verhindert stressinduzierte Transkriptionsänderungen. Ohne intakte Zellen den strengen Dissoziationsschritten auszusetzen, SNRNA-Seq-Probenahme bewahrt die Integrität der zum Zeitpunkt der Dissoziation vorhandenen Transkripte besser bei.
Wie minimiert snrna-seq falsche Genexpressionsmuster?
Dies minimiert das Auftreten von falschen Genexpressionsmustern, die durch den Dissoziationsprozess selbst verursacht werden. Reife Ribosomen, die ausschließlich in zytoplasmatischen Kompartimenten gefunden wurden. Ihre nachgeschalteten Transkriptionsziele bleiben somit unverändert. Vergleichende Studien, bei denen beide Techniken auf demselben Gewebe angewendet werden.
Wie können kurze Lysedauern dazu beitragen, transkriptomische Verzerrungen in snrna-seq zu kontrollieren?
Zusätzliche Faktoren wie kurze Lysedauern helfen bei der Kontrolle transkriptomischer Verzerrungen. Eine längere Exposition auf Eis vor dem Abfahren mit der Vorbereitung der Einzelnukleus-Bibliothek kann sich negativ auf die Datenqualität auswirken, indem Transkriptionsänderungen initiiert werden. Sorgfältige Timing -Ergänzung solcher Effekte. Ausreichende anfängliche Gewebemengen sorgen auch für hochwertige Kernenpräparate, die für eine robuste Profilierung bei methodischen Einschränkungen erforderlich sind.
Wie minimieren optimierte snRNA-seq-Workflows technische Artefakte?
Gesamt, Optimierte snRNA-seq-Workflows minimieren präanalytische Faktoren, die die biologische Interpretation komplizieren. Ihre sanfte Extraktion des Kerngehalts zeigt Genexpressionsprofile, die nach technischen Artefakten weniger geprägt sind als endogene biologische Prozesse. Dies verleiht mehr Klarheit und Vertrauen für Entdeckungen, die in Bezug, seltene Subpopulationen, und zelluläre Spezialisierungsprogramme in komplexen In -vivo -Systemen.
Wie können Forscher snrna-seq anwenden, um das biologische Verständnis voranzutreiben?
Als innovative Technik, die unsere Sicht auf die zelluläre Komplexität verfeinert, SNRNA-seq öffnet neue Grenzen für verschiedene Forschungsanwendungen. Einige Beispiele unterstreichen das Potenzial, wissenschaftliche Fortschritte voranzutreiben:
- Entwicklungsbiologie: Der Vergleich der Kerne aus unterschiedlichen embryonalen Stadien könnte stadienspezifische regulatorische Programme leiten, die die Differenzierung leiten. Räumlich aufgelöste snrna-seq-Mapping-Entwicklungsgradienten können Strukturmechanismen aufdecken.
- Krankheitspathologie: Das Profilieren von Erkrankungen im Vergleich zu gesunden Gewebekern kann pathologische assoziierte Veränderungen in seltenen Zelltypen identifizieren, die Krankheitsmechanismen beleuchten. Patient -Probenanalysen könnten Präzisionsdiagnostik und Therapeutika beeinflussen.
- Neuropsychiatrie: Die Untersuchung des archivierten Post-Mortem-Gehirngewebes über snrna-seq charakterisiert die Neurodiversität der psychiatrischen Bedingungen. Längsschnittstudien können Längsbiomarker und therapeutische Ziele aufdecken.
- Evolutionsstudien: SNRNA-seq macht praktikable Kreuzungsvergleiche, die bisher aufgrund der Unterschiede zwischen den Organismen in den Gewebeeigenschaften eine Herausforderung darstellen. Solch „Phylo-snrna-seq“ informiert unser Verständnis von konservierten und unterschiedlichen Regulierungsprozessen.
- Multi-OMICS-Integration: Kombination von snrna-seq mit räumlichen Profilerstellungstechniken, die räumlich aufgelöste epigenomische und transkriptomische Karten ergeben. Die Integration räumlicher Transkriptdaten in proteomische oder metabolomische Schichten bietet einen Kontext auf Systemebene auf.
Zusammenfassung
Als eine von mehreren aufstrebenden Techniken revolutionieren die Einzelzellanalyse, SNRNA-Seq-Katalysen-Entdeckungen, indem bisher unlösbare Proben experimentell verfolgbar gemacht werden. Die Skalierung von nuklearen Omics verspricht neue Erkenntnisse, die verschiedene Bereiche von der Regenerativmedizin bis zur Präzisionsonkologie vorantreiben. Zusammen mit komplementären Ansätzen, SNRNA-seq hilft dabei.