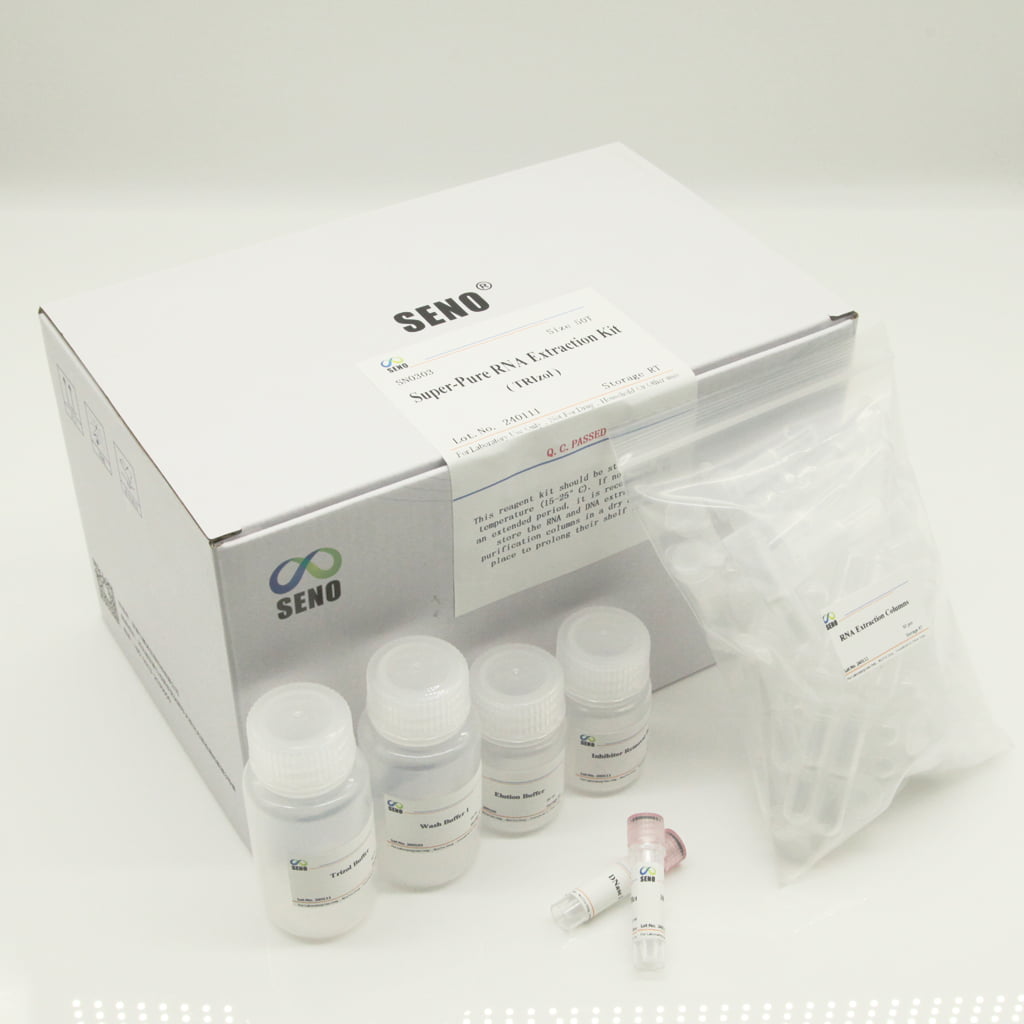
- Bestandteile des Reagenzienkits
| Spezifikationen | 50T | 100T |
| Katze. NEIN. | Sn0303 | SN0304 |
| RNA -Extraktionssäulen (Satz) | 50 (Satz) | 100 (Satz) |
| DNase I | 1ml | 1ml |
| 10 × Reaktionspuffer | 1 ml | 2 × 1 ml |
| Trizol -Puffer | 50 ml | 2 × 50 ml |
| Inhibitorentfernungspuffer | 30 ml | 2 × 30 ml |
| Waschpuffer 1 | 15 ml | 2 × 15 ml |
| Elutionspuffer | 20 ml | 2 × 20 ml |
| Bedienungsanleitung | 1 | 1 |
- Lagerung
Dieses Reagenzienkit sollte bei Raumtemperatur gelagert werden (15-25℃) in einer trockenen Umgebung und ist stabil für 12 Monate. DNase I enthält einen Konservierungsmittel, ermöglicht den Transport bei Raumtemperatur, aber zur Langzeitlagerung, es sollte bei -20℃ gehalten werden.
- Anweisungen zur Verwendung des Reagenzienkits
3.1 Dieses Kit ist für molekularbiologische Forschungszwecke bestimmt und sollte nicht zur Diagnose oder Behandlung von Krankheiten verwendet werden.
3.2 Einige Bestandteile des Kits enthalten Reizstoffe; Es ist ratsam, die erforderlichen Vorsichtsmaßnahmen zu treffen (wie das Tragen von Schutzkleidung und Schutzbrillen).
3.3 Die Verwendung dieses Kits erfordert zusätzliche Ausrüstung wie eine Hochgeschwindigkeitszentrifuge, Wasserbad (Metallbad), Vortexmixer, Wasserfreies Ethanol, flüssiger Stickstoff, Chloroform, steriles entionisiertes Wasser, und EP-Röhren.
- Einführung in das Reagenzienkit
Dieses RNA -Reinigungskit verwendet das herkömmliche Trizol in Kombination mit Säulenmembranmethode zur schnellen Reinigung der Pflanze, Tier, Gewebe, Zelle, mikrobielle RNA, und Pilzhyphen -RNA. Es ist für die meisten Arten geeignet. Dieses RNA -Reinigungskit kann auf überschrittene Pflanzengewebe angewendet werden 100 mg. Die mit diesem Kit extrahierte RNA hat einen extrem niedrigen DNA -Gehalt. Wenn die Empfindlichkeit gegenüber DNA in Experimenten ein Problem darstellt, Es wird empfohlen, die DNase I -Verdauung auf der Spalte zu verwenden.
Das RNA -schnelle Reinigungskit kann die gesamte RNA aus Proben extrahieren (einschließlich nuklearer RNA und zytoplasmatischer RNA) innerhalb 1 Stunde. Die extrahierte RNA kann direkt für RT verwendet werden-PCR, Northern Blot, und andere Anwendungen.
- Experimentelle Prinzipien und Verfahren

- Extraktionsprozess
Vorsichtsmaßnahmen vor Beginn des Experiments:
A. Vor Gebrauch, Fügen Sie die angegebene Menge an absolutem Ethanol hinzu WaschenPuffer 1 Nach dem Etikett auf der Reagenzienflasche, und aktivieren Sie das Kontrollkästchen auf dem Etikett, um anzuzeigen, dass absolutes Ethanol hinzugefügt wurde.
B. Elutionspuffer ist ein 0.1x TE-Lösung mit minimalem EDTA-Gehalt. Wenn EDTA nachfolgende Experimente beeinflusst, Es wird empfohlen, den Elutionspuffer durch steriles entionisiertes Wasser zu ersetzen.
- Probenverarbeitung:
A. Gewebe: Wenn frische Materialien nicht sofort verwendet werden können, Legen Sie sie in flüssiges Stickstoff und lagern Sie sie bei -80 ° C.. Getrocknete Materialien können bei Raumtemperatur gelagert werden. Mahlen Sie 30 ~ 80 mg Gewebe in flüssigem Stickstoff und fügen Sie hinzu 1 ML Trizol Puffer zur Homogenisierung.
B. Monoschicht kultivierte Zellen: Das Kulturmedium anhalten und hinzufügen 1 ML Trizol Puffer zur Homogenisierung.
C. Zellsuspension: Zentrifuge, um Zellen zu sammeln und hinzufügen 1 ML Trizol Puffer zur Homogenisierung.
2. Nach dem Hinzufügen Trizol -Puffer zur Probe, durch Pipettieren gründlich mischen, Ermöglichen Sie eine vollständige Probenlyse, und bei Raumtemperatur inkubieren für 5 Protokoll.
3. Chloroform in ein Verhältnis von hinzufügen 200 μl per 1 Ml Trizol -Puffer, kräftig schütteln 30 Sekunden, und bei Raumtemperatur inkubieren für 2 Protokoll.
4. Zentrifugen Sie das Lysat bei 4 ° C, 12,000 U/min für 10 Protokoll. RNA wird in der oberen wässrigen Phase vorhanden sein.
5. Übertragen Sie den Überstand vorsichtig in ein neues Zentrifugenröhrchen, Vermeiden Sie die Sammlung der mittleren und unteren Schichten. (Notiz: Etwa 400 Es können μl Flüssigkeit übertragen werden, das kann für einige Arten weniger sein.)
6. Fügen Sie ein gleiches Volumen des vorgekalten absoluten Ethanols hinzu, schnell mischen. (Zum Beispiel, hinzufügen 400 μl Lysat, hinzufügen 400 μl von absolutem Ethanol. Wenn das Lysatvolumen kleiner ist als 400 μl, Reduzieren Sie proportional die Menge an absolutem Ethanol. Ein leichter Niederschlag kann sich nach Zugabe von Ethanol bilden, Es wirkt sich jedoch nicht auf die nachfolgenden Experimente aus.)
7. Übertragen Sie die erhaltene Flüssigkeit in eine RNA -Reinigungssäule (etwa 650-700 μl pro Zeit), Zentrifuge um mehr als 8,000 U/min für 1 Minute, Entsorgen Sie den gesammelten Abfall, und setzen Sie das Sammelröhrchen für den nächsten Schritt wieder in die Reinigungssäule ein.
8. Schritt wiederholen 7, Hinzufügen der verbleibenden Flüssigkeit zur RNA -Reinigungssäule, Zentrifuge um mehr als 8,000 U/min für 1 Minute, Verwerfen Sie den Abfall und das Sammelrohr.
9. Platzieren Sie die RNA -Reinigungssäule in ein neues Sammelrohr, hinzufügen 300 μl des Inhibitorentfernungspuffers, Zentrifuge um mehr als 8,000 U/min für 1 Minute, Entsorgen Sie den Abfall, und stöbern Sie für den nächsten Schritt die RNA -Reinigungssäule in das Röhrchen ein.
10. Hinzufügen 80 μl von DNase I.Arbeitslösung für die RNA -Reinigungssäule, bei 20 ° C bis 30 ° C inkubieren für 15 Protokoll. (Bereiten Sie die DNase I -Arbeitslösung vor: 62 μl rnase-freies Wasser, 8 μl 10 × Reaktionspuffer, Und 10 μl dnase i, wieder gut machen 80 μl DNase I Arbeitslösung.)
11. Hinzufügen 300 μl des Inhibitorentfernungspufferszur RNA -Reinigungsspalte, Zentrifuge um mehr als 8,000 U/min für 1 Minute, Entsorgen Sie den Abfall, und stöbern Sie für den nächsten Schritt die RNA -Reinigungssäule in das Röhrchen ein.
12. Hinzufügen 700 μl Waschpuffer 1zur RNA -Reinigungsspalte, Zentrifuge bei 14,000 U/min (20,000×g) für 2 Protokoll, Erweitern Sie die Zentrifugationszeit bei Bedarf für eine trockenere Membran. (Notiz: Bestätigen Sie die Zugabe von Ethanol zum Waschenpuffer 1; Das Präsenz von Ethanol beeinflusst nachfolgende Experimente signifikant. Stellen Sie sicher, dass die Membran nach der Zentrifugation vor der Elution trocken ist. Entsorgen Sie den Abfall und das Sammelröhrchen. Nach der Verwendung des Waschpuffer 1, Die Membran an der RNA -Reinigungssäule sollte nur eine leichte Farbe haben. Entfernen Sie die RNA -Reinigungssäule nach der Zentrifugation vorsichtig, Stellen Sie sicher.)
13. Platzieren Sie die RNA -Reinigungssäule in eine neue Zentrifugenröhre, tropfen 100 μl Elutionspufferauf die Membran, bei Raumtemperatur inkubieren für 5 Protokoll (15° C bis 25 ° C.), und Zentrifuge um mehr als 8,000 U/min für 1 Minute. (Notiz: Elusionsrna mit 50 μl Elutionspuffer kann die RNA -Konzentration erhöhen, aber die Gesamt -RNA -Ausbeute verringern.)
14. Wiederholen Sie den vorherigen Schritt. (Notiz: Mit einem neuen Zentrifugenröhrchen kann die RNA zum zweiten Mal sammeln oder das ursprüngliche Sammelrohr fortsetzen, um RNA zu sammeln.)








Rezensionen
Es gibt noch keine Rezensionen.