1. Bestandteile des Reagenzienkits
| Spezifikationen | 50T | 100T |
| Katze. NEIN. | Sn0241 | SN0242 |
| DNA-Extraktionssäulen (Satz) | 50 (Satz) | 100 (Satz) |
| Huminsäure Entfernung Reagenz i | 50ml | 2x50ml |
| Huminsäure -Entfernungsreagenz II | 50ml | 2x50ml |
| Reagenzpuffer ⅳ | 30 ml | 2×30 ml |
| Reagenzpuffer C Plus | 30 ml | 2×30 ml |
| Inhibitorentfernungspuffer | 30ml | 2x30ml |
| Lysozym | 1ml | 2x1ml |
| Proteinase K | 1ml | 2x1ml |
| RNase A | 1ml | 2x1ml |
| Waschpuffer 1 | 15 ml | 2 × 15 ml |
| Elutionspuffer | 20 ml | 2 × 20 ml |
| Bedienungsanleitung | 1 | 1 |
2. Lagerung
Dieses Reagenzienkit sollte bei Raumtemperatur gelagert werden (15-25℃) und bei trockenen Bedingungen, mit einer Haltbarkeit von 12 Monate. Die DNA-Extraktions-Reinigungssäulen können aufbewahrt werden 1 Jahr in einer kühlen und trockenen Umgebung. Lysozym, Proteinase K, Und RNase A enthalten Konservierungsstoffe, ermöglicht den Transport bei Raumtemperatur, aber zur Langzeitlagerung, Sie sollten bei -20℃ aufbewahrt werden.
3. Anweisungen zur Verwendung des Reagenzienkits
3.1 Dieses Reagenzienkit ist für die molekularbiologische Forschung bestimmt und sollte nicht zur Diagnose oder Behandlung von Krankheiten verwendet werden.
3.2 Einige Komponenten im Reagenzienkit enthalten Reizstoffe. Schutzmaßnahmen wie das Tragen von Schutzkleidung und Schutzbrille werden empfohlen.
3.3 Während der Verwendung dieses Reagenzienkits, eine Hochgeschwindigkeitszentrifuge, Wasserbad (Metallbad), Vortexmixer, Wasserfreies Ethanol, steriles entionisiertes Wasser, und EP-Röhren müssen vom Benutzer vorbereitet werden.
4. Einführung in das Reagenzienkit
Dieses Kit bietet eine schnelle und effiziente Methode zur Reinigung mikrobieller genomischer DNA aus dem Boden, Kot, und Erbrochene Proben. Es wird häufig für die mikrobielle DNA -Extraktion aus dem Boden verwendet, Kot, und Erbrechen.
Dieses Kit entfernt effektiv Probenverunreinigungen und Inhibitoren, Reduzierung der Hemmreaktionen in nachgeschalteten Experimenten wie z. PCR. Der gesamte Reinigungsprozess erfordert keine giftigen Reagenzien wie Phenolchloroform. Die extrahierte DNA kann direkt für PCR verwendet werden, Southern Blot, und andere Anwendungen.
5. Experimentelle Prinzipien und Verfahren
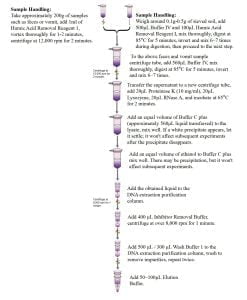
6. Extraktionsprozess
Vor Beginn des Experiments:
A. Reagenzpuffer Iv kann bei niedrigen Temperaturen ausfallen. Wir empfehlen das Erhitzen bei 65 ℃ für 5 Protokoll. Nachdem sich der Niederschlag aufgelöst hat, es kann normal verwendet werden.
B. Vor Gebrauch, Fügen Sie die angegebene Menge an wasserfreiem Ethanol hinzu Waschpuffer 1 Wie auf dem Flaschenetikett angegeben. Markieren Sie auf dem Etikett ein Häkchen, um die Zugabe von wasserfreiem Ethanol anzuzeigen.
C. Elutionspuffer ist ein 0.1x TE-Lösung mit minimalen Mengen an EDTA enthält. Wenn EDTA nachfolgende Experimente beeinflussen könnte, Es ist ratsam, den Elutionspuffer durch steriles entionisiertes Wasser zu ersetzen.
- Probenverarbeitung (Inhibitorentfernung):
A. Fäkal- und Erbrochene Proben: Fügen Sie ungefähr 200 mg der zu extrahierenden Probe hinzu, hinzufügen 1ml Huminsäureentfernungsreagenz i i, Vortex gründlich für 1-2 Protokoll, Zentrifuge bei 12,000 U/min für 2 Protokoll, Den Überstand verwerfen, hinzufügen 560μl von Puffer IV zum Pellet, gründlich umkehren und mischen, verdauen Sie bei 85 ℃ für 5 Protokoll, umdrehen und mischen 6-7 Zeiten während der Verdauung, Dann fahren Sie mit Schritt 2.
B. Bodenproben: Wiegen Sie ungefähr 0,1 g-0.5G von gesiebten Boden, hinzufügen 500μl von Puffer IV und 100 & mgr; l Huminsäure -Entfernungsreagenz I I., gründlich umkehren und mischen, verdauen Sie bei 85 ℃ für 5 Protokoll, umdrehen und mischen 6-7 Zeiten während der Verdauung, Dann fahren Sie mit Schritt 2.
(Notiz: Huminsäure Entfernung Reagenz I/Huminsäure Entfernung Reagenz II wird hauptsächlich für Böden mit hohem Huminsäuregehalt verwendet, wie Waldböden und Sedimentböden. Hinzufügen 1ml Huminsäureentfernungsreagenz i i zur Probe, Wirbel, schütteln für 1-2 Protokoll, Zentrifuge bei 8,000 U/min für 2 Protokoll, Den Überstand verwerfen, dann hinzufügen 1ml Huminsäure -Entfernungsreagenz II II, Wirbel, Zentrifuge bei 8,000 U/min für 2 Protokoll, Den Überstand verwerfen. Dieser Schritt kann die Huminsäure -Inhibitoren von Boden weiter reduzieren, aber die DNA -Ausbeute signifikant reduzieren.)
- Zentrifuge bei 12,000 U/min für 2 Protokoll, Übertragen Sie den Überstand in eine neue Zentrifugenröhre (ca. 500 μl Flüssigkeit übertragen), Fügen Sie 20 μl Proteinase K hinzu (10 mg/ml), 20μl Lysozym, und 20 μl RNase a, und inkubieren bei 65 ℃ für 2 Protokoll.
- Fügen Sie eine gleiche Menge hinzu Reagens Puffer C Plus (ca. 560 μl Flüssigkeit übertragen) zum Lysat, gut mischen. Wenn ein weißer Niederschlag erscheint, Lass es sich niederlassen; Es wird nach dem Verschwinden des Niederschlags nach dem Verschwinden des Niederschlags keine Auswirkungen auf die nachfolgenden Experimente beeinflussen.
- Fügen Sie ein gleiches Volumen von Ethanol hinzu zu Reagens Puffer C Plus (z.B., hinzufügen 560μl von Reagens Puffer C Plus, Dann 560 μl Ethanol hinzufügen), gut mischen. Es kann Niederschläge geben, Es wird sich jedoch nicht auf die nachfolgenden Experimente auswirken.
- Fügen Sie die erhaltene Flüssigkeit zur DNA -Extraktionsreinigungssäule hinzu (Ärmel) (jeweils etwa 650–700 μl), Zentrifuge bei über 8,000 U/min für 1 Minute, Entsorgen Sie die gesammelte Abfallflüssigkeit, und stöbern Sie das Sammelrohr in die DNA -Extraktionsreinigungssäule ein (Ärmel) für den nächsten Schritt.
- Hinzufügen 400μl -Inhibitorentfernungspuffer, Zentrifuge bei über 8,000 U/min für 1 Minute, Entsorgen Sie die Abfallflüssigkeit, und wieder einversetzen DNA-Reinigung Säule in die Hülse für den nächsten Schritt.
- Hinzufügen 500μl Waschpuffer 1 zur DNA-Extraktions-Reinigungssäule (Ärmel), Zentrifuge bei 14,000 U/min (20,000×g) für 1 Minute.
- Hinzufügen 300μl Waschpuffer 1 Wieder zur DNA -Extraktionsreinigungsspalte (Ärmel), Zentrifuge bei 14,000 U/min (20,000×g) für 2 Protokoll.
- Platzieren Sie die DNA-Extraktions-Reinigungssäule (Ärmel) in ein neues Zentrifugenröhrchen füllen, Öffne den Deckel, und inkubieren bei 65 ℃ für 2 Protokoll. Dieser Schritt kann bei Bedarf erweitert werden, um Ethanol so weit wie möglich zu verdampfen, um zu verhindern, dass Ethanolreste nachgeschaltete Experimente beeinflussen.
- Tropfen 100μl Elutionspuffer auf die Membran, Zentrifuge bei 12,000 U/min für 2 Protokoll.
(Notiz: 1. Die Eluting -DNA mit einem Elutionspuffer von 50 μl kann die DNA -Konzentration erhöhen, aber die GesamtdNA -Ausbeute verringern; 2. Die eluierte DNA kann erneut auf die DNA-Extraktions-Reinigungssäule aufgetragen werden, wieder zentrifugiert bei 12,000 U/min für 2 Protokoll, und gesammelt, um die DNA -Ausbeute zu erhöhen.)







Rezensionen
Es gibt noch keine Rezensionen.